por
Claire G. Williams and James L. Hamrick
Department of Forest Sciences
Texas A&M University, USA
RESUMEN
La utilización de pequeñas poblaciones para mejora genética en generaciones avanzadas, seleccionadas a base de rasgos de adaptación y selviculturales, ha suscitado algunas reservas sobre la suficiencia de la diversidad genética. Se emplearon marcadores moleculares para medir la diversidad genética en rodales naturales de Pinus taeda y en poblaciones domesticadas para mejora genética a fin de resolver dos cuestiones: 1) ¿Son las selecciones élite, descendientes de tercera generación, menos diversas genéticamente que sus progenitores de primera generación o de rodales naturales?, 2) ¿Es superior la diversidad genética con una estrategia de mejora genética de poblaciones múltiples, en comparación con una estrategia de mejora genética de carácter jerárquico pero similar a la de rodales naturales?
La mayor parte de la diversidad alélica de los rodales naturales de P. taeda se encontraba en las poblaciones domesticadas. La variabilidad genética se concentraba análogamente en las selecciones de tercera y primera generación, independientemente del progreso generacional. La subdivisión de la población de mejora genética, preferentemente acompañada de la estrategia de poblaciones múltiples, equilibró el avance genético a corto plazo y la flexibilidad genética a largo plazo. Se tratan brevemente las aplicaciones a otras especies de árboles forestales.
INTRODUCCION
El equilibrio entre el avance genético y la diversidad genética es una meta conveniente de los programas a largo plazo de mejora genética de árboles. Pocos de estos programas han alcanzado ya la etapa de domesticación en la que constituye un problema la pérdida de diversidad debida a la mejora genética; la mayoría de ellos han avanzado menos de cuatro ciclos de mejora. La prevalencia de las pequeñas poblaciones puede acelerar la pérdida de diversidad genética en las poblaciones domesticadas. Cabe esperar que las poblaciones más pequeñas sean propensas a la pérdida de alelos debida a la selección, a la mayor endogamia y a la deriva genética.
El Pinus taeda L. es una especie pionera monoica de polinización anemófila que se regenera naturalmente es rodales coetáneos a lo largo de la región costera del Atlántico y en las tierras bajas de todo el sur de los Estados Unidos. Está clasificada en la subsección Australes, que incluye el Pinus caribaea. La longevidad y el transporte a largas distancias de la semilla y el polen, motivan que este pino tenga dentro de las poblaciones unos niveles sumamente elevados de diversidad genética (Hamrick et al. 1992).
Las poblaciones de mejora genética de generación avanzada se subdividen para controlar la tasa de endogamia, para permitir la selección en cuanto a diferentes series de rasgos y para acentuar la mejora de las selecciones mejores (p. ej. Williams y Hamrick 1995). Estas poblaciones de élite pequeñas y aisladas mediante el control de la genealogía, se apartan extraordinariamente de las grandes poblaciones naturales de cruzamiento lejano, con dispersión a gran distancia del polen y la semilla. Los criterios de diversidad genética deben reflejar esta deriva en el tamaño de la población y en el flujo genético.
Los mejoradores que están a favor de subdivisión deben elegir entre dos soluciones generales frente a la pérdida de alelos al azar. La primera solución es el sistema jerárquico en el que se infunde periódicamente un germoplasma no emparentado en una población élite. Es jerárquico porque la población de mejora genética se subdivide verticalmente de acuerdo con el valor genético del germoplasma. Los sistemas jerárquicos se han definido en conjunto como la estrategia de conservación genética HOPE (Eriksson et al. 1994).
La segunda solución consiste en subdividir basándose más en la ascendencia común que en el valor genético. Una población de mejora puede dividirse en repeticiones o subpoblaciones que representan diferentes orígenes o planes de selección. La pérdida de alelos al azar se produce en cualquier replicación pero como promedio las frecuencias de los genes permanecen bastante constantes entre una generación y la siguiente. Esta es la base de las estrategias de mejora genética mediante poblaciones múltiples (MPBS) y mediante subdivisión de poblaciones en la mejora genética de árboles forestales (Burdon y Namkoong 1983; Namkoong 1984; Eriksson et al. 1994; Cotterill 1984; Lowe y van Buijtenen 1986).
Predicciones de los niveles de diversidad genética utilizando la estrategia de mejora genética HOPE
El HOPE se refiere al Sistema de Mejora Genética de Carácter Jerárquico y Abierto diseñado en la Universidad de Guelph para el maíz (Kannenberg 1984). Los programas HOPE se caracterizan por un equilibrio entre el avance a corto plazo y el mantenimiento a largo plazo de la diversidad genética. Cada programa se compone de una jerarquía de poblaciones de mejora con un nivel sucesivamente superior de resultados, que culmina en una población élite de la cual se extraen selecciones a utilizar en la producción comercial.
El nivel inferior de la jerarquía HOPE lo constituyen los “archivos de conservación de genes”. Estos consisten en una subpoblación de bajo costo para conservar alelos que están presentes en las primeras generaciones de mejora genética de árboles. El próximo nivel jerárquico lo constituye la subpoblación principal donde se introducen complejos genéticos nuevos en las líneas élites. El nivel máximo de la jerarquía es una subpoblación élite que rápidamente se ciclaría para conseguir un avance genético máximo por unidad de tiempo. En nuestra población HOPE hay únicamente dos tercios: la subpoblación media y la élite. Es de suponer que la alternativa HOPE de dos escalas tendrá menores niveles de diversidad genética que la estrategia de poblaciones múltiples.
Predicción de los niveles de diversidad genética utilizando una estrategia de poblaciones múltiples (MPBS)
La estrategia de mejora genética con poblaciones múltiples (MPBS) combina el avance a corto plazo, el incremento de germoplasma y los objetivos de conservación genética dentro de la misma población de mejora (Namkoong, 1984; Barnes 1984; Eriksson et al. 1994). Los sistemas de mejora con poblaciones múltiples son aplicables a especies de las que todavía se dispone de gran número de alelos. Bajo la MPBS, se pueden establecer pequeñas subpoblaciones procedentes de diferentes orígenes de la semilla y, dentro de cada población, se utilizan programas de selección independientes. En la práctica, un plan MPBS tiende a tener un conjunto continuo de subpoblaciones. Algunas se seleccionan para diversas metas en cuanto a productos, mientras que otras son replicaciones, compartiendo un origen regional común y una meta en cuanto a productos (Burdon y Namkoong 1983). Los diversos orígenes y metas de selección distinguen la mejora con poblaciones múltiples de otras estrategias de mejora como las estrategias de núcleo o de subdivisión de poblaciones. Esto contrasta con el modelo HOPE en el que el avance a corto plazo, el incremento de germoplasma y los objetivos de conservación genética se cumplen con subpoblaciones independientes.
Orígenes de las poblaciones para los programas HOPE y MPBS
Todos los genotipos de este estudio se muestrearon dentro del litoral atlántico de la parte oriental de los EUA desde los 32°N, 80°30'W a los 38°N, 77°30'W. Para los rodales naturales, se seleccionaron al azar 48 árboles, muestreados en rodales naturales en Andrews, Carolina del Sur; Washington, Carolina del Norte y Windsor, Virginia. Se asignaron selecciones de generación avanzada a dos programas diferentes de mejora y conservación, antes de los análisis electroforéticos para 19 loci isoenzimáticos. Se incluyeron selecciones de primera, segunda y tercera generación como parte de dos estrategias mutuamente excluyentes de mejora y conservación. La estrategia de poblaciones múltiples consistió en seis subpoblaciones no emparentadas (MPBS). La segunda, una estrategia jerárquica (HOPE) consistió en dos subpoblaciones, una población élite y una subpoblación principal de selecciones hacia atrás no emparentadas. Existe un solape entre ambas opciones; algunas selecciones de la subpoblación principal HOPE estaban también representadas en varias subpoblaciones MPBS.
RESULTADOS Y DISCUSION
La mayor parte de la diversidad genética (medida mediante isoenzimas), encontrada en los rodales naturales estaba aún presente en las poblaciones en las que se había realizado la selección. Esto era especialmente cierto para las poblaciones de mejora MPBS y HOPE. Las subpoblaciones de tercera y primera generación tenían niveles análogos de diversidad genética en cuanto a los rasgos moleculares incluidos en el estudio y presentaban sólo una ligera disminución de diversidad genética de tales rasgos en relación con los rodales naturales. Existía todavía la mayor parte de la diversidad genética dentro de cada subpoblación, más que entre subpoblaciones, independientemente de los ciclos de generación de la selección y mejora genética.
La pérdida de alelos en las características moleculares consideradas se limitó a los alelos de menor frecuencia (q < 15%). Había también algunos alelos de baja frecuencia (q < 15%) en las poblaciones seleccionadas que no estaban presentes en los rodales naturales. Esto no fue inesperado ya que los rodales naturales eran muestras locales más que progenitores de selecciones reales. Si se hubieran muestreado más rodales naturales dentro de la región, se habrían muestreado también más alelos de baja frecuencia. El nivel de diversidad genética dentro de tres rodales naturales podría haber estado representado defectivamente en relación con la población seleccionada. Este puede ser el caso de las selecciones originales de primera generación que se muestrearon a partir de rodales individuales, esto es, 34 selecciones de primera generación representan 34 rodales diferentes.
Diversidad genética de los MPBS y HOPE en comparación con los rodales naturales
Cuando se mide mediante marcadores moleculares, las poblaciones agregadas de mejora en el MPBS y el HOPE conservaban más diversidad genética que la población élite sola (ELITE). La diversidad genética en el MPBS era elevada aunque el tamaño de la población de mejora MPBS tenía números efectivos menores (que variaba del 15 al 100% del número censado) para las subpoblaciones.
La suma de poblaciones múltiples (MPBS) tenía una diversidad genética, a nivel de isoenzimas, comparable con los rodales naturales (Cuadro 1) aunque sus tamaños efectivos de población eran inferiores (Nev = 5 a 22) a los de los rodales naturales (Cuadro 1). La heterozigosidad media esperada era inferior para los MPBS (Ne=0,180) que para los rodales naturales (He=0,213) pero las diferencias de los valores no eran identificables. Los promedios de alelos por locus (A) eran equivalentes; para los loci polimórficos (Apoly), el MPBS tenía un número comparable de alelos por locus en relación con la población natural. La población total MPBS capturaba casi tantos alelos en total (50) como el muestreo de los tres rodales naturales (52) (Cuadro 1); el número de loci polimórficos cambiaba del 93 al 90% en relación con los rodales naturales. La opción MPBS presentaba el máximo nivel de diversidad genética para un tamaño efectivo dado de población.
La opción HOPE se componía de menos selecciones (N = 90) que la MPBS pero tenía mayores tamaños efectivos de población (Nev = 40–41) (Cuadro 1). Había menos loci polimórficos (85%) y el número promedio de alelos por locus era inferior que en el MPBS o en las poblaciones naturales (Cuadro 1). La mayor parte de la diversidad genética de los rodales naturales se encontraba aún en el HOPE. Sin embargo, en la estrategia HOPE estaba representado un número total menor de alelos (47).
Existen todavía algunas dudas sobre si deben añadirse subpoblaciones adicionales. El peligro real del MBPS para un programa de una sola especie está en añadir demasiadas subpoblaciones. Había suficiente diversidad genética con seis (o menos) subpoblaciones. Las subpoblaciones adicionales extraídas de la periferia del ámbito de la especie pueden no incrementar en este momento la diversidad genética de esta población MPBS. La inseguridad de los productos, la adaptabilidad a largo plazo y la demanda de reforestación son criterios apropiados para justificar subpoblaciones adicionales ya que los niveles de diversidad genética son suficientemente elevados por el momento.
La afirmación de que estas subpoblaciones MPBS contienen “suficiente” diversidad genética debe matizarse con dos supuestos. En primer lugar, hemos supuesto que los alelos marcadores raros (frecuencia genética q < 10 %) no son importantes para un avance genético o una adaptabilidad a largo plazo. Lo esencial del problema de la conservación forestal radica en la importancia de estos alelos marcadores raros existiendo poco acuerdo sobre esta cuestión. Una escuela teórica sostiene que los raros alelos contribuyen poco al valor global de adaptabilidad, que aparecen en gran parte como mutaciones desfavorables y pueden ser relictos evolutivos (Lindgren y Gregorius 1976; Brown 1989). Una opinión contraria es que la pérdida de alelos raros puede comprometer la flexibilidad a largo plazo para resistir futuras plagas y atender a posibles cambios climáticos futuros y a ciertas metas de domesticación. Análogamente, estas pérdidas podrían indicar una pérdida de alelos en los loci que cifran la adaptabilidad a corto plazo o rasgos importantes económicamente no considerados en un programa determinado.
En segundo lugar, las estimaciones de diversidad genética se aplican únicamente a la población de mejora. La diversidad genética de las poblaciones de mejora se traduce en una diversidad genética suficiente en el esfuerzo de reforestación (es decir, huertos semilleros o plantaciones forestales) sólo si las selecciones de cada subpoblación están igualmente representadas en los huertos semilleros y en las plantaciones. Este es un primer paso para medir la diversidad genética en poblaciones domesticadas de árboles forestales. El próximo paso es determinar si los marcadores seleccionados al azar representan la diversidad genética del genoma del P. taeda de forma apropiada para el éxito o fracaso de la mejora genética de los árboles.
Cuadro 1. Estimaciones de la diversidad genética del Pinus taeda L. para la opción de poblaciones múltiples (MPBS) y para la opción HOPE. El MPBS corresponde a una estrategia de mejora con poblaciones múltiples, el HOPE a una estrategia de mejora piramidal y el Elite a una población élite de mejora de tercera generación manejada para un avance a corto plazo. Heterozigosidad media esperada (H), porcentaje de loci polimórficos (% P), número de alelos por locus (A) y número de alelos por loci polimórficos (Apoly). Gen indica el nivel generacional de la subpoblación; los programas de mejora del Pinus taeda no han utilizado históricamente generaciones solapadas (datos procedentes de Williams et al. 1995).
| Opción/subpoblación | Gen | Ho | He | % P | A | Apoly |
| NAT - VA | 0 | 0,199 (0,10) | 0,210 (0,034) | 89 | 2,21 | 2,35 |
| NAT - NC | 0 | 0,186 (0,10) | 0,213 (0,034) | 95 | 2,32 | 2,39 |
| NAT - SC | 0 | 0,185 (0,10) | 0,210 (0,034) | 95 | 2,32 | 2,39 |
| Total NAT | - | - | 0,213 | 100 | 2,63 | 2,63 |
| MPBS / ELA | 3 | 0,168 (0,015) | 0,183 (0,043) | 79 | 2,05 | 2,33 |
| MPBS / ELB | 3 | 0,176 (0,017) | 0,185 (0,045) | 68 | 1,89 | 2,31 |
| MPBS / HD | 2 | 0,152 (0,014) | 0,174 (0,045) | 63 | 1,89 | 2,42 |
| MPBS / NCS | 1 | 0,152 (0,018) | 0,171 (0,042) | 84 | 2,05 | 2,25 |
| MPBS / SCS | 1 | 0,182 (0,023) | 0,198 (0,051) | 68 | 1,95 | 2,38 |
| MPBS / GGC | 2 | 0,200 (0,030) | 0,204 (0,036) | 79 | 1,84 | 2,07 |
| Total MPBS | - | - | 0,180 | 90 | 2,50 | 2,67 |
| HOPE/Elite | 3 | 0,171 (0,012) | 0,185 (0,045) | 75 | 2,05 | 2,40 |
| HOPE/Main | 1 | 0,164 (0,013) | 0,195 (0,045) | 89 | 2,21 | 2,35 |
| Total HOPE | - | - | 0,181 | 85 | 2,40 | 2,65 |
Diversidad genética de las selecciones élite, de tercera generación
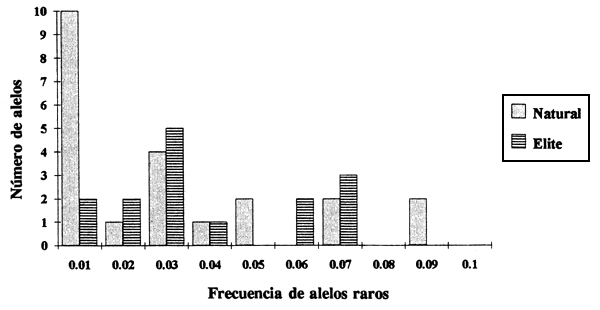
Las selecciones élite de tercera generación tenían niveles similares de diversidad genética al de otras poblaciones seleccionadas, independientemente de las diferencias generacionales. Las selecciones élite de tercera generación presentaban una disminución de la diversidad genética, en relación con los rodales naturales, en los marcadores de isoenzimas considerados, pero esto no era detectable estadísticamente (Cuadro 1). La subpoblación élite tenía menos diversidad genética que los conjuntos del MPBS o del HOPE (Cuadro 1) y presentaba una mayor pérdida de alelos. La subpoblación élite sola tenía un total de 41 alelos en comparación con los 50 del MPBS, 47 del HOPE y 52 de la población natural. La distribución de alelos raros variaba en más en la población seleccionada de tercera generación; la pérdida de alelos pareció ser máxima, del orden de 0,01 (Figura 1).
En este estudio, el método de muestreo genético era más importante que el número de generaciones de domesticación. No había una tendencia asociada con el nivel generacional. Por ejemplo, las subpoblaciones NCS y SCS estaban compuestas ambas de selecciones de primera generación aunque la SCS tenía únicamente 68% de loci polimórficos en comparación con el 84% de loci polimórficos para la NCS (Cuadro 1). Los valores para la SCS y la NCS se solapaban con la población de tercera generación (Cuadro 1).
Figura 1. Distribución de alelos raros en el pino taeda natural y en selecciones élite de tercera generación (datos de Williams et al. 1995)
Aplicación a otras especies de árboles forestales
Las coníferas de distribución amplia tienden a tener pequeñas diferencias entre poblaciones con respecto a los marcadores moleculares (Hamrick et al. 1992; Millar y Libby 1992). La mayoría de los alelos son comunes a todas las poblaciones y pocos son localizados o son raros. Se necesitarán relativamente pocas selecciones para representar adecuadamente la variación alélica en los marcadores moleculares a nivel de especie. Estos resultados serán aplicables ampliamente a otras especies de árboles forestales como los eucaliptos, algunas coníferas y álamos, que tienen todos ellos un modelo similar de variación alélica (p. ej. Prober y Brown 1994) y si la serie inicial de selecciones representan una muestra de extensión amplia.
REFERENCIAS
Barnes, R.D. (1984). A multiple population breeding strategy for Zimbabwe. In: Barnes R.D. and G.L.Gibson (eds.) Proceedings IUFRO Provenance and genetic improvement strategies in tropical forest trees. Commonwealth Forestry Institute, Oxford UK.
Brown, A.H.D. (1989). Core collections: a practical approach to genetic resources management. Genome 31: 818–824.
Burdon, R.D. & Namkoong, G. (1983). Short Note: Multiple populations and sublines. Silvae Genetica 32 (5/6): 221–222.
Cotterill, P.P. (1984). A plan for breeding radiata pine. Silvae Genetica 33(2/3): 84–90.
Eriksson, G.; Namkoong, G. & Roberds, J.H. (1994). Dynamic gene conservation for uncertain futures. Forest Ecology and Management 62: 15–37.
Hamrick, J.L.; Godt, M.J.W. & Sherman-Broyles, S.L. (1992). Factors affecting levels of genetic diversity in woody plant species. New Forests 6: 95–124.
Kannenberg, L.W. (1984). Utilization of genetic diversity in crop breeding. In: Yeatman C.W., Kafton D., Wilkes G. (eds) Plant genetic resources: a conservation imperative, AAAS Selected Symposium 87 edn. Westview Press, Boulder, CO
Lowe, W.J. & van Buijtenen, J.P. (1986). The development of a sublining system in an operational tree improvement program. In: Proceedings IUFRO Conference on Breeding Theory, Progeny testing and Seed Orchards, Williamsburg, VA.
Millar, C.I. & Libby, W.J. (1992). Strategies for conserving clinal, ecotypic and disjunct population diversity in widespread species. pp. 149–170. In Genetics and Conservation of Rare Plants (D. Falk and K. Holsinger, eds). Oxford Press, New York. 283 p.
Namkoong, G. (1984) A control concept of gene conservation. Silvae Genetica 33 (4/5):160–163.
Prober, S.M. & Brown, A.H.D. (1994). Conservation of the grassy white box woodlands: population genetics and fragmentation of Eucalyptus albens. Conservation Biology 8(4): 1003–1013.
Williams, C.G. and Hamrick, J.L. (1995). Elite populations for conifer breeding and gene conservation. Canadian Journal Forest Research (in press).
Williams, C.G.; Hamrick, J.L. & Lewis, P.O. (1995). Multiple-population versus hierarchical conifer breeding programs: A comparison of genetic diversity levels. Theoretical and Applied Genetics 90: 584–594.
Recursos Genéticos Forestales No. 23. FAO, Roma (1995)
Manuscrito recibido en abril de 1995