par
Gunter M. Rothe
Institut für Allgemeine Botanik, Fachbereich Biologie
Johannes Gutenberg Universität
Saarstr. 21, D-6500 Mainz, Allemagne
1. INTRODUCTION
Des études sur la génétique des arbres forestiers ont été effectuées en utilisant principalement trois catégories de caractères héréditaires simples: marqueurs morphologiques, variantes de monoterpènes, et isoenzymes.
Les caractères morphologiques sont aisés à évaluer, mais leur utilisation dans les études génétiques est limitée, du fait que les caractéristiques morphologiques phénotypiques sont généralement déterminées par plusieurs gènes, et que les facteurs quantitatifs sont fortement influencés par le milieu. Par conséquent, on ne parvient pas à distinguer la contribution respective des différents gènes, et les différences génétiques précises entre individus ne sont pas décelables.
Chez les gymnospermes, des gènes uniques déterminent la teneur de certains monoterpènes. Toutefois, l'interprétation génétique de la répartition des monoterpènes est complexe, et l'analyse d'un grand nombre d'échantillons par chromatographie en phase gazeuse-liquide est longue et coûteuse.
Par comparaison, les isoenzymes sont d'emploi relativement aisé. On a commencé à les utiliser en génétique forestière il y a une quinzaine d'années, et elles sont maintenant largement appliquées; leur emploi se fonde sur l'hypothèse qu'elles sont le produit direct d'allèles spécifiques. Une fois qu'elles sont identifiées, on dispose de méthodes satistiques pour étudier leur distribution (fréquences) à l'intérieur d'une population ou entre populations.
2. LES ALLELOENZYMES, MARQUEURS GENETIQUES
2.1 Les organismes diploïdes à reproduction sexuée reçoivent de chacun des parents un ensemble complet de gènes. En conséquence, ils possèdent chaque gène en double; les gènes homologues sont appelés allèles. Si l'on trouve plus d'un allèle au locus d'un gène structurel, les diverses substances enzymatiques qu'ils produisent peuvent apparaître soit dans un seul organisme diploïde (hétérozygote), soit dans différents membres (homozygotes) d'une espèce.
Le nombre d'allèles a un locus donné dépend de l'espèce et du locus lui-même. Les gènes sont qualifiés de polymorphes s'ils comprennent deux ou plusieurs allèles. Les gènes invariants (monomorphes) ne se prêtent pas aux études génétiques.
Pour interpréter correctement les schémas d'isoenzymes2, il faut connaître leur composition en sous-unités (nombre de polypeptides formant la molécule complète d'enzyme) (Fig. 1).
Les gènes qui constituent le code des alléloenzymes sont identifiés par une abréviation du nom de l'enzyme, par exemple “Aat” pour aspartate amino-transférase. Si un gène comprend plus d'un locus (emplacement précisément défini sur la molécule d'ADN), ces loci sont indiqués par une lettre majuscule (ou un chiffre) placée en suffixe au nom du gène, exemple: Aat-A, Aat-B (ou Aat-1, Aat-2). Les allèles correspondants peuvent être indiqués par des chiffres consécutifs, par exemple: Aat-A1, Aat-A2, etc. (ou Aat-11, Aat-12) (Figures 2, 3; Tableaux 1, 2).
Les alléloenzymes peuvent être utilisées dans les études génétiques si les conditions suivantes sont remplies:
Le génotype est exprimé phénotypiquement par des schémas ou spectres d'enzymes donnés, qui peuvent être observés au laboratoire. Une condition préalable est que les formes d'enzymes intéressantes montrent une ségrégation mendélienne (tel qu'indiqué en Fig.1);
Au moins un locus par enzyme est polymorphe.
Les génotypes spécifiques peuvent être identifiés par un des moyens suivants:
Examen de la descendance provenant de croisements contrôlés;
Etudes comparatives de tissus haploïdes et diploïdes d'un individu (endosperme haploïde et embryon diploïde des gymnospermes, ou pollen haploïde et embryon diploïde des angiospermes);
Application de la loi de Hardy-Weinberg dans des modèles théoriques (Rothe, 1988).
2.2 MOYENS DE DÉTECTION DES ISOENZYMES
L'identification des isoenzymes est basée sur leur spécificité élevée vis-à-vis du substrat et leurs différences de mobilité électrophorétique. Si deux variantes alléliques d'une enzyme diffèrent dans le nombre des aminoacides chargés, leurs différences de charge nette peuvent être mises à profit pour les séparer par électrophorèse. La structure primaire (chaîne peptidique) de ces enzymes est codée dans les deux brins homologues d'ADN dans l'individu, et en conséquence les gènes alléliques peuvent être identifiés par des alléloenzymes de charges différentes. Malheureusement, toutefois, les deux tiers des aminoacides protéinogènes n'ont pas de charge électrique, ce qui réduit les possibilités d'identification des variantes alléliques d'enzymes existantes à moins de 30 pour cent du nombre total présent. En revanche, pour les variantes qui ont une charge électrique, les méthodes électrophorétiques sont relativement simples à appliquer en pratique. Il suffit de placer des extraits bruts de cellules ou de tissus sur un support de séparation électrophorétique tel que gel d'amidon-, cellogelR- ou gel de polyacrylamide, et ensuite de teinter le gel à l'aide d'un colorant qui permet seulement à une enzyme déterminée et à ses multiples formes moléculaires de devenir visibles, tout en laissant non colorées les autres protéines (et enzymes) séparées (Rothe & Maurer, 1986; Kephart, 1990). Finalement les formes d'enzymes colorées sont comptées, et les fréquences estimées utilisées pour les calculs génétiques ultérieurs (voir section 2. ci-dessous; cf également Figures 2, 3).
3. APPLICATION DE LA RECHERCHE SUR LES ALLELOENZYMES
3.1. IDENTIFICATION DE LOTS DE SEMENCES
Les différences de fréquences des alléloenzymes peuvent être utilisées pour déterminer l'origine d'un lot de semences ou de plants provenant d'un verger à graines ou de différentes populations d'une essence forestière donnée (Adams, 1983 et Bergmann, 1975a. Voir aussi Houston, 1978, pour un exemple d'identification de six sources géographiquement différentes de semences de Quercus rubra par utilisation des peroxydases). Cependant, en particulier en Europe Centrale, l'identification des provenances d'épicéa commun (Picea abies) est malaisée, du fait que l'on a introduit un grand nombre de provenances et que leur origine réelle est inconnue (Bergmann, 1975a).
Pour l'identification des provenances d'épicéa, il faut analyser au moins 7 graines par arbre sur 200 arbres pour déterminer les fréquences d'alléloenzymes (Adams, 1983; Bergmann, 1975a). Selon Bergmann (1975a), on a trouvé des fréquences d'alléloenzymes identiques dans des graines, qu'elles proviennent d'individus d'une certaine population, ou d'un mélange de graines de cette population particulière. L'examen de 100 échantillons de génomes diploïdes récoltés dans un verger à graines (ou de 50 endospermes haploïdes d'un conifère) permet de détecter les allèles se trouvant à une fréquence de 0,03 ou plus, avec une probabilité de 95% (Adams, 1983). Toutefois, si l'on soupçonne une sérieuse contamination par du pollen provenant de sources extérieures au verger et que l'on recourt à l'analyse d'iosenzymes pour le démontrer, il est recommandé de doubler l'échantillon ci-dessus, et de prendre 100 endospermes haploïdes de graines (Adams, 1983).
Dans de nombreux pays on a divisé les forêts géographiquement, topographiquement et écologiquement en zones de semences. Chaque zone est présumée représenter une unité écologique relativement homogène, à l'intérieur de laquelle les génotypes, en principe, sont adaptés pour être utilisés dans n'importe quelle autre partie de la même zone, tandis que les semences provenant de l'extérieur de la zone sont a priori moins bien adaptées (Adams, 1983). En restreignant les semences utilisées en reboisement à celles récoltées dans les zones de semences choisies, on espère ainsi assurer qu'elles seront bien adaptées aux conditions de la station de plantation. L'identité des lots de semences peut être vérifiée après leur récolte en utilisant des techniques d'isoenzymes. Toutefois, si la diversité génétique entre populations est faible (do < 0,10)3, cela restreindra l'emploi des études d'isoenzymes pour l'identification des lots de semences provenant de diverses zones de semences (Adams, 1983).
3.2 IDENTIFICATION DE CLONES
Les isoenzymes peuvent aussi être utilisées pour identifier des arbres étroitement apparentés, ou des clones (Adams, 1983). Les études d'isoenzymes peuvent se faire sur des bourgeons hivernaux, des feuilles ou des aiguilles de l'année. Pour l'identification de clones il suffit de comparer les schémas phénotypiques d'isoenzymes, même sans connaître leur base génétique (Cheliak & Pitel, 1984; Bergmann, 1987). Dans une étude, l'analyse de six ou sept systèmes variables d'enzymes, comprenant 10 à 20 loci différents, a permis la certification de 50 à 100 clones d'une variété multi-clonale d'épicéa commun (Picea abies) (voir Tableau 3). Cette application des études d'isoenzymes a une grande importance potentielle, du fait que les variétés multiclonales ont été développées dans certains pays dans le cadre d'une stratégie contribuant à éviter la diminution de la diversité génétique dans les générations futures de populations artificielles (Rothe, 1990).
3.3 DÉTERMINATION DE LA FIABILITÉ DES CROISEMENTS ET DU TAUX D'AUTOFÉCONDATION
La fiabilité des croisements contrôlés peut être évaluée en examinant la distribution des isoenzymes dans les graines résultant de ces croisements, et en comparant les résultats avec ceux que l'on peut théoriquement attendre en se basant sur les génotypes des parents (Adams, 1983). Une pollinisation contrôlée peut être considérée comme un échec si les graines d'un échantillon sur quatre (ou d'un sur deux pour les conifères) contiennent des allèles qui ne sont présents dans aucun des deux parents supposés (Adams, 1983). Des expérimentations menées sur le douglas (Pseudotsuga menziesii) ont montré que la plupart des échecs, dans les croisements contrôlés, sont dus à la contamination par un pollen étranger (Adams, 1983).
D'autre part, on peut utiliser les isoenzymes comme marqueurs génétiques pour identifier les arbres autofécondés dans les vergers à graines (Müller, 1976; Moran, Bell & Matheson, 1980; Shen, Rudin & Lindgren, 1981). Etant donné que l'autofécondation influe de manière égale sur tout le génome, on peut la révéler en étudiant un seul locus polymorphe. Chez l'épicéa commun (Picea abies), au locus leucine aminopeptidase B (Lap-B), on a constaté une autofécondation pour deux génotypes sur quatre. Le taux d'autofécondation a été estimé à 15% (Miller, 1976). Etant donné que la diversité génétique des semences d'arbres autofécondés est réduite, il est généralement désirable d'identifier et éliminer de tels individus des vergers à graines (Shaw & Allard, 1981).
3.4 IDENTIFICATION DES ESPECES D'ARBRES
La variation de la distribution d'alléloenzymes peut être utilisée pour identifier des espèces ou des hybrides différents. Les graines de deux espèces de mélèzes, Larix decidua et Larix kaempferi, ont pu être distinguées par analyse de la mobilité de l'enzyme shikimate déshydrogénase. Chez L. kaempferi, l'enzyme migre plus rapidement vers l'anode que chez L. decidua. Chez des hybrides de ces deux espèces, les deux variantes d'enzymes ont pu être identifiées (Bergmann, 1975a).
Les chênes (Quercus spp) constituent un groupe taxonomique complexe. On en trouve aux Etats-Unis plus d'une cinquantaine d'espèces, et le genre comprend au total plus de 300 espèces. Certaines possèdent des caractères morphologiques et anatomiques aisément identifiables, mais d'autres sont difficiles à identifier et à différencier entre elles. L'identification taxonomique est en outre compliquée par la présence d'hybridation entre un certain nombre d'espèces. Plus de 60 hybrides ont été signalés rien qu'aux Etats-Unis (Fernald & Gray, 1950).
L'étude de bourgeons et de tissus de cotylédons de glands provenant de diverses sources de semences de chêne rouge (Quercus rubra L.), chêne des teinturiers (Q. velutina Lam.), chêne blanc (Q. alba L.), chêne bicolore (Q. bicolor Willd), chêne à gros fruits (Q. macrocarpa Michx.) et chêne à chinquapin (Q. muehlenbergii Engelm.) ont montré que la plupart des brins d'isoenzymes de leucine aminopeptidase, alcool déshydrogénase, phosphatase acide et aspartate transaminase sont communs à deux ou plusieurs de ces espèces (Tobolski, 1978). Cette similarité dans la distribution des isoenzymes traduit sans doute leur étroite parenté taxonomique - et par conséquent génétique. Toutefois, des individus de certaines espèces ont montré à l'analyse des génotypes nettement distincts; ceux-ci pouvaient également être aisément distingués par leurs caractères phénotypiques (Tobolski, 1978).
Les espèces Quercus petraea (chêne rouvre) et Q. robur (chêne pédonculé) ont pu être classifiées en fonction de leurs distributions de peroxydase (Olsson, 1975). Certaines populations de Q. petraea montrent des signes d'introgression avec Q. robur.
3.5 CARACTÉRISTIQUES DES POPULATIONS D'ARBRES FORESTIERS
Outre les emplois mentionnés ci-dessus, les caractéristiques génétiques suivantes de populations ont été étudiées par les fréquences d'alléloenzymes:
degré de variabilité génétique à l'intérieur d'une population;
variation entre populations;
facteurs pouvant rendre compte de la variation observée.
La structure génétique des populations, se traduisant par la variabilité entre loci dans les études d'alléloenzymes, est soumise aux forces évolutionnaires telles que mutation, sélection et dérive génétique aléatoire. La variabilité entre loci, identifiable dans les études de laboratoire, dépend de la taille de l'échantillon et des fréquences de gènes au locus étudié (Nei & Roychoudhury, 1974). On a trouvé dans les populations d'arbres forestiers des degrés d'hétérozygotie4 (H) variant entre 0,19 et 0,41, les valeurs les plus élevées étant caractéristiques des conifères (Tableaux 4 et 5).
La variation allélique peut être définie comme étant “la proportion attendue d'hétérozygotes par locus et par population”, et elle est généralement désignée par la lettre h. Les calculs de valeurs moyennes de h dans dix populations différentes d'épicéa (épinette) de Sitka (Picea sitchensis (Boug.) Carr.), importante essence forestière de la côte ouest d'Amérique du Nord, ont fourni des valeurs variant entre 0,475 (glucose 6-phosphate déshydrogénase, G6pdh) et 0,001 (phosphoglucomutase, Pgm-2). Cette ample variation de h, avec des valeurs proches de zéro, est typique de diverses espèces de conifères (Yeh & El-Kassaby, 1980).
Le degré de diversité génétique chez les conifères est généralement élevé. Cela est dû: (i) à une sélection diversifiante pour l'adaptation macrogéographique, (ii) à une sélection uniformisante pour la différenciation microgéographique, et (iii) à un système de reproduction qui favorise le mouvement de gènes à l'intérieur de sous-populations et entre elles. L'hétérosis peut contribuer elle aussi au maintien de la variabilité génétique (Yeh & El-Kassaby, 1980). Des études de variation génétique dans dix populations IUFRO5 d'épicéa de Sitka (Picea sitchensis) ont montré que: (i) le degré de polymorphisme varie considérablement d'un locus à un autre; (ii) plusieurs populations étaient identiques dans leur degré et leur modèle de variabilité génétique, en ce qui concerne la plupart des loci étudiés; alors que (iii) certains loci déterminés montrent de grandes différences entre populations (Yeh & El-Kassaby, 1980). En dépit de l'éloignement géographique entre les sources de semences étudiées, les différences dans la constitution génétique de l'épicéa de Sitka ne s'accroissent pas de manière appréciable en fonction de la distance.
Des différences génétiques entre populations les exposent différemment à la sélection naturelle au niveau de la population, favorisant la sélection de populations (Yeh & El-Kassaby, 1980). Cette conclusion a été confirmée pour diverses autres essences forestières, telles que Picea abies (L.) Karst. (Bergmann, 1974), Pinus monticola Dougl. (Steinhoff, Joyce & Fins, 1983) et Pseudotsuga menziesii (Mirb.) Franco (Yeh & O'Malley, 1980). En Scandinavie, les fréquences d'allèles situés au locus de la phosphatase-B acide étaient moins élevées dans les populations méridionales d'épicéa commun que dans les populations septentrionales (Bergmann, 1978).
D'une manière générale, les conifères montrent des degrés élevés d'hétérozygotie. Une exception notable à cette règle est le pin rouge, Pinus resinosa (Fowler & Morris, 1977). L'une des théories pour expliquer la faible variabilité de Pinus resinosa est qu'elle serait le résultat d'un sérieux rétrécissement de population qui s'est probablement produit au pléistocène, où par suite de la glaciation la répartition de l'espèce s'est trouvée réduite à de petites populations dans des aires refuges.
La variabilité génétique chez les arbres à feuilles caduques est généralement plus faible que chez les conifères. Dans 17 populations de Quercus, on a trouvé une proportion moyenne de loci polymorphes de 30%, avec 2,32 allèles par locus polymorphe. Les faibles estimations de diversité génétique entre populations indiquent sans doute qu'il existe une variabilité génétique plus grande à l'intérieur des populations qu'entre elles (Manos & Fairbrothers, 1987).
3.6 ADAPTATION DES ARBRES A DIFFÉRENTS MILIEUX
Les arbres sont des organismes qui ont une longue durée de vie, et doivent par conséquent être bien adaptés à leurs habitats naturels et aux fluctuations des conditions écologiques qui peuvent intervenir sur une longue période. Ils y parviennent soit par une adaptation générale à l'intérieur de la population, soit par une adaptation spécifique de la population à des conditions de station spécifiques. Chez l'épicéa commun (Picea abies) et le sapin de Douglas (Pseudotsuga menziesii), on a identifié un locus codant la phosphatase acide, qui montre une variation significative liée au milieu. Chez les deux espèces, les allèles Acp codant des enzymes à simple bande dominent dans les parties les plus froides de l'aire de répartition de l'espèce, tandis que les allèles codant des enzymes à double bande se rencontrent plus fréquemment dans toutes les zones de climat plus tempéré (Bergmann, 1975).
La variation entre populations peut être mesurée par un chiffre unique, la distance génétique (do) (voir par exemple Nei & Roychoudhury, 1974, et Surles, Hawrick & Bongarten, 1989). Des populations totalement différentes génétiquement se caractérisent par une valeur de do égale à 1, tandis que cette valeur est de zéro pour des populations identiques. Dans des peuplements finlandais de Picea abies, on a observé une distribution totalement aléatoire des gènes à l'intérieur des peuplements en utilisant des alléloenzymes de leucine aminopeptidase (Lap), de peroxydase (Pod) et d'estérase (Est) (Tigerstedt, 1973). Toutefois, pour un certain nombre d'autres espèces, il semble exister une corrélation entre distance génétique et éloignement géographique, par exemple chez Camellia japonica L. (Wendel & Parks, 1985), Picea abies (L.) Karst. (Bergmann, 1975), Pinus monticola Dougl. (Steinhoff, Joyce & Fins, 1983) et Pseudotsuga menziesii (Mirb.) Franco (Yeh & O'Malley, 1980). L'isolement par la distance peut être un important facteur dans la différenciation des populations de ces espèces (cf Perry & Knowles, 1988).
3.7 INFLUENCE DE LA POLLUTION ATMOSPHÉRIQUE SUR LA STRUCTURE GÉNÉTIQUE DES ARBRES FORESTIERS
De nombreuses données concernant la différenciation génétique des arbres relativement à leur sensibilité à la pollution ont été passées en revue par Gerhold (1977). Des différences statistiquement significatives existent entre populations d'épicéa commun (Picea abies) concernant leur sensibilité au SO2. Les populations les plus tolérantes semblent être celles d'origine septentrionale d'altitude (Tzschacksch & Weiss, 1972). L'hétérozygotie moyenne d'individus d'épicéa poussant dans le massif du Harz (Allemagne), qui n'avaient pas souffert d'une exposition à SO2 plus O3, s'est révélée plus élevée que celle d'individus qui avaient été endommagés (Bergmann & Scholz, 1987). Toutefois, des provenances d'épicéa tolérantes à SO2 ont montré des fréquences plus faibles d'allèles rares se trouvant aux loci Gpdh-A1 et Mdh-C3 que des sous-populations sensibles à SO2 (Bergmann & Scholz, 1987). Contrairement aux variables complexes du milieu, les polluants atmosphériques simples tels que SO2 ou O3 entraînent une sélection dans le sens d'une réduction de la diversité génétique (Bergmann & Scholz, 1987). Des observations similaires ont été faites pour le hêtre (Fagus sylvatica), chez lequel la sélection de génotypes résistants s'est faite principalement à la première génération (F1) (Müller-Starck, 1985).
Des études de l'influence des polluants atmosphériques sur la structure génétique des arbres forestiers on peut tirer deux conclusions principales:
Des arbres fortement hétérozygotes semblent être mieux adaptés à une multiplicité d'impacts du milieu agissant simultanément que des arbres à faible degré d'hétérozygotie;
La contamination par les polluants atmosphériques provoque une diminution de la diversité génétique de la population. Les raisons de cette diminution de la diversité (et par conséquent de l'adaptabilité générale) sont (1) des niveaux accrus de sélection directionnelle et (2) une circulation de gènes diminuée.
On suppose que la résistance des arbres forestiers aux polluants atmosphériques est déterminée par des gènes préadaptatifs. Ce sont des gènes hautement adaptatifs, qui existent déjà dans le génome, mais qui ne sont pas identifiables avant exposition à l'agent toxique (Gregorius, Hattemer, Bergmann & Müller-Starck, 1985).
Note de l'éditeur: Pour plus d'information sur la biotechnologie dans les programmes d'amélioration des arbres, voir également l'article de Cheliak, W.M. & Rogers, D.L., dans la section (iv) de Publications récentes intéressantes.
Figure 1.
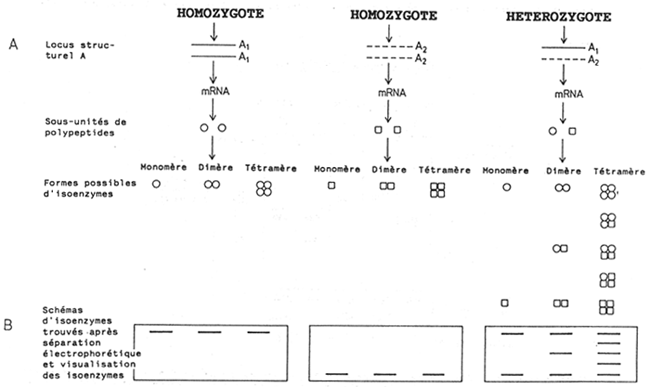
Représentation schématique des spectres d'isoenzymes donnant naissance à des hétérozygotes lorsque l'enzyme est un mono-, di- ou tétramère (comprenant un, deux ou quatre polypeptides) et qu'au moins deux allèles différents apparaissent au locus qui les code. On suppose en outre que les sous-unités se combinent librement et présentent une ségrégation mendélienne.
Figure 2.
Quercus robur L.
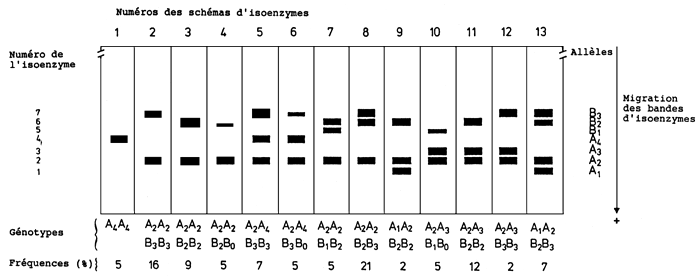
Schémas d'isoenzymes de leucine aminopeptidase trouvés chez Quercus robur, et interprétation génétique. Les isoenzymes ont été extraites de 35 tissus de cotylédons de glands de Q. robur, séparées par électrophorése avec gel de polyacrylamide, et enfin visualisées dans le gel par une méthode de coloration histochimique. Les glands provenaient d'un peuplement de Q. robur du Palatinat rhénan (Allemagne). Au total 13 schémas différents d'isoenzymes ont été observés, comprenant sept isoenzymes différentes. On suppose que deux loci différents (Lap-A et Lap-B) codent ces sept isoenzymes, représentant quatre allèles au locus A et trois allèles au locus B (Dankwardt & Rothe, non publié).
Figure 3.
Quercus petraea (Matt.) Liebl.
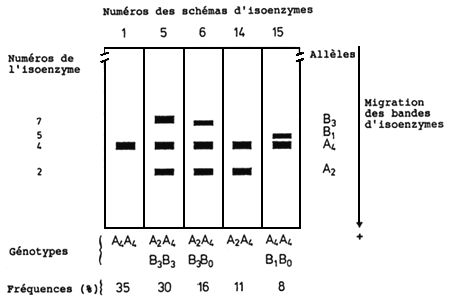
Schémas d'isoenzymes observés sur 35 tissus de cotylédons de glands de Quercus petraea. Les glands provenaient d'un peuplement du Palatinat rhénan (Allemagne). Par comparaison avec Q. robur (Fig. 2), on n'a identifié que cinq schémas d'isoenzymes au lieu de 13. Les schémas 1, 5 et 6 sont identiques à ceux de même numéro chez Q. robur, mais les fréquences diffèrent. Les schémas 14 et 15 ont été trouvés exclusivement chez Q. petraea, tandis que les schémas 2 à 4 et 7 à 13 sont typiques de Q. robur. En conséquence les populations de ces deux espèces peuvent être distinguées par leurs schémas d'isoenzymes. Toutefois, l'identification d'individus des deux espèces exige une étude plus poussée de leurs systèmes d'isoenzymes (Dankwardt & Rothe, non publié).
Tableau 1
Loci de gènes d'enzymes et nombres d'allèles trouves chez l'épicéa commun (Picea abies (L.) Karst.) et utilisés pour les études génétiques
| Aspartate aminotransferase | Aat-A | Aat-B | Aat-C | ||
| 3 | 3 | 1 | Lagercrantz 1988 | ||
| 4 | 4 | 4 | Lundquist 1979 | ||
| 2 | 1 | 3 | Poulsen et al. 1983 | ||
| 2 | 3 | Muona et al. 1987 | |||
| Acid phosphatase | Acp-A | Acp-B | |||
| 1 | 4 | Bergmann 1978 | |||
| 7 | Lunquist 1977 | ||||
| Diaphorase | Dia-A | Dia-B | |||
| 2 | 1 | Lagercrantz 1988 | |||
| Esterase | Est-A | Est-B | Est-C | ||
| 3 | 3 | 2 | Bergmann 1973 | ||
| Glucose-6-phosphate dehydrogenase | Gpdh-A | ||||
| 2 | Schoiz & Bergmann 1984 | ||||
| 3 | Muona et al. 1987 | ||||
| Glutamate dehydrogenase | Gdh-A | Gdh-B | |||
| 2 | Scholz & Bergmann 1984 | ||||
| 2 | Lundquist 1979 | ||||
| 2 | Lagercrantz 1988 | ||||
| Glucose-6-phosphate dehydrogenase | Gpi-A | Gpi-B | |||
| 5 | 1 | Lagercrantz 1988 | |||
| Isocitrate dehydrogenase | Idh-A | Idh-B | |||
| 2 | 2 | Scholz & Bergmann 1984 | |||
| 3 | 1 | Lagercrantz 1988 | |||
| Leucine aminopeptidase | Lap-A | Lap-B | |||
| 1 | 4 | Bergmann 1973 | |||
| 4 | 4 | Lundquist 1979 | |||
| 3 | 4 | Poulsen et al. 1983 | |||
| 5 | 5 | Lagercrantz 1988 | |||
| Malate dehydrogenase | Mdh-A | Mdh-B | Mdh-C | Mdh-D | |
| 1 | 2 | 2 | 2 | Poulsen et al. 1983 | |
| 4 | Scholz & Bergmann 1984 | ||||
| ? | 2 | 1 | ? | Lagercrantz 1988 | |
| Phosphoglucose isomerase | Pgi-A | Pgi-B | |||
| ? | 4 | Muona et al. 1987 | |||
| 6-Phosphogluconate dehydrogenase | Pgd-A | Pgd-B | Pgd-C | ||
| 2 | 4 | 3 | Poulsen et al. 1983 | ||
| 4 | 1 | 1 | Lagercrantz 1988 | ||
| Phosphoglucomutase | Pgm-A | Pgm-B | |||
| 2 | 2 | Poulsen et al. 1983 | |||
| 2 | 2 | Scholz & Bergmann 1984 | |||
| Superoxide dismutase | Sod-A | Sod-B | |||
| 1 | 1 | Lagercrantz 1988 |
Tableau 2
Loci de gènes d'enzymes et nombres d'allèles trouvés chez des arbres à feuilles caduques et utilisés pour les études génétiques
| Espèce | Loci d'enzymes et nombre d'allèles | Référence | ||||||||||||||||||||||||||||||||||
| Aat- | Aco- | Acp- | Ald- | Est- | Gdh | Gpdh- | Idh- | Lap- | Mdh- | Per- | Pgd- | Pgi- | Pgm- | |||||||||||||||||||||||
| A | B | C | A | B | A | B | A | B | A | B | A | B | A | B | A | B | A | B | C | A | B | C | D | A | B | C | D | A | B | C | A | B | C | |||
| Alnus crispa | 1 | 1 | 2 | 2 | 1 | 1 | 1 | 1 | 2 | ? | 2 | 3 | 1 | 2 | 3 | 1 | Bousquet et al. 1987 | |||||||||||||||||||
| Alnus rugosa | ? | 2 | 2 | 1 | 1 | 1 | 1 | 1 | 4 | 2 | 4 | 3 | 1 | 2 | Bousquet et al. 1987 | |||||||||||||||||||||
| Castanea sativa | 2 | 3 | 2 | Fineschi 1988 | ||||||||||||||||||||||||||||||||
| Fagus sylvatica | 2 | 4 | 4 | 2 (3) | 2 | 4 | 4 | 4 | ? | 3 | 2 | 2 (4) | 3 | 2 | Kim 1979, Müller-Starck 1985 | |||||||||||||||||||||
| Juglans regia | 2 | 2 | 1 | ? | 2 | 1 | 2 | 3 | 1 | Arulsekar et al. 1985 | ||||||||||||||||||||||||||
| Populus balsamifera | 3 | 2 | 2 | 2 | 2 | 1 | 3 | 1 | 3 | 1 | 4 | Farmer et al. 1988 | ||||||||||||||||||||||||
| Populus tremula | 3 | 2 | 1 | 2 | 2 | 2 | Guzina 1978, Bergmann 1987 | |||||||||||||||||||||||||||||
| Populus tremuloides | 1 | 1 | 3 | 1 (4) | 2 (3) | 3 | 2 | 3 | 4 | 2 | 3 | 3 | 4 | 2 | 2 | 3 (1 | 4 3) | 3 | 3 | 4 | Hyun et al. 1987 Cheliak and Pitel 1984 | |||||||||||||||
| Populus trichocarpa | 2 | 1 | 4 | 3 | 1 | 2 | 1 | 2 | 2 | 1 | 2 | 1 | 2 | Weber and Stettler 1981 | ||||||||||||||||||||||
| Robinia pseudoacacia | 1 | 3 | 3 | 3 | 3 | 4 | 5 | 4 | 3 | 4 | 3 | 3 | 3 | 3 | 3 | 2 | 3 | 4 | 3 | Surles et al. 1988 | ||||||||||||||||
| Quercus petraea | 1 (2) | 2 | 2 | 1 | 3 | 3 | 9 | 4 | Dankwardt and Rothe unpubl. Aas 1987 | |||||||||||||||||||||||||||
| Quercus robur | 1 (2) | 2 | 2 | 1 | 3 | 4 | 9 | 4 | Dankwardt and Rothe unpubl. Aas 1987 | |||||||||||||||||||||||||||
| Quercus subg. Erythrobalanus | ? | 4 | 3 | ? | ? | 2 | 3 | 1 | 3 | 1 | 2 | ? | 8 | 5 | 4 | Manos and Fairbrothers 1987 | ||||||||||||||||||||
Sigles:
Aat: aspartate aminotransferase
Aco: aconitase
Acp: acid phosphatase
Ald: aldolase
Est: esterase
Gdh: glutamate dehydrogenase
Gpdh: glucose-6-phosphate dehydrogenase
Idh: isocitrate dehydrogenase
Lap: leucine aminopeptidase
Mdh: malate dehydrogenase
Per: peroxidase
Pgd: 6-phosphogluconate dehydrogenase
Pgi: phosphoglucose isomerase
Pgm: phosphoglucomutase
Tableau 3
Caractérisation de 16 clones d'épicéa (Picea abies [L.] Karst.) à débourrage tradif par l'étude des schémas d'isoenzymes de six enzymes sur les aiguilles de l'année (Rothe, 1990)
| Numéro du clone: | 1G | 7G | 8G | 10G | 14G | 15G | 16G | 17G | 18G | 21G | 84B | 89B | 93B | 94B | 95B | 105B |
| Schémas phénotypiques d'isoenzymes | ||||||||||||||||
| ADH | 1 | 2 | 7 | 3 | 7 | 5 | 3 | 5 | 7 | 5 | 8 | 8 | 1 | 6 | 4 | 8 |
| EST | 4 | 4 | 5 | 5 | 5 | 5 | 2 | 1 | 2 | 3 | 1 | 1 | 1 | 1 | 1 | 1 |
| G6pDH | 3 | 1 | 1 | 3 | 3 | 3 | 2 | 3 | 3 | 3 | 3 | 3 | 6 | 4 | 5 | 4 |
| LDH | 1 | 1 | 2 | 1 | 2 | 2 | 1 | 1 | 1 | 1 | 2 | 2 | 1 | 2 | 2 | 1 |
| 6PGDH | 3 | 1 | 4 | 4 | 2 | 2 | 3 | 5 | 5 | 6 | 3 | 3 | 3 | 7 | 5 | 3 |
| SHDH | 4 | 4 | 3 | 4 | 3 | 3 | 1 | 1 | 2 | 2 | 3 | 3 | 2 | 2 | 2 | 3 |
Sigles:
ADH = alcool déshydrogénase;
EST = estérase (colorée par α-naphtylphosphate et Fast blue RR);
G6pDH = glucose-6-phosphate déshydrogénase;
LDH = lactate déshydrogénase;
6PGDH = 6-phosphogluconate déshydrogénase;
SHDH = shikimate déshydrogénase
NB: Les clones numéros 1 à 24 ont été sélectionnés à partir d'arbres de la provenance Goldap (G), tandis que les clones numéros 84 à 105 ont été sélectionnés à partir d'épicéas de la provenance Borken (B), les deux étant situées en Pologne.
Tableau 4
Hétérozygotie moyenne (H) de quelques populations d'arbres à feuilles caduques (cf Perry & Knowles, 1988)
| Espèce | H |
| Acer saccharum | 0.27 |
| Alnus crispa | 0.136 |
| Populus balsamifera | 0.037 |
| Populus tremuloides | 0.42 |
| Populus trichocarpa | 0.09 |
| Quercus subg. Erythrobalanus | 0.081 |
Tableau 5
Variabilité génétique de populations de conifères, exprimée par l'hétérozygotie moyenne (H) (cf Kormutak et al., 1982)
| Espèce | H |
| Pinus contorta | 0.19 |
| Pinus jeffreyi | 0.26 |
| Pinus lambertina | 0.26 |
| Pinus silvestris | 0.41 |
| Pinus taeda | 0.34 |
| Pseudotsuga menziesii | 0.33 |
| Sitka spruce | 0.15 |
REFERENCES
Aas, G. 1988 Untersuchung zur Trennung und Kreuzbarkeit von Stiel- und Traubeneiche (Quercus robur L. und Q. petraea (Matt.) Liebl.), Inaugural-Dissertation zur Erlangung der Doktorwürde der Forstwissenschaftlichen Fakultät der Ludwig-Maximilians Universität, München.
Adams, W.T. 1983 Application of isozymes in tree breeding, in: Tanksley, S.D. and T.J. Orton (Eds.): “Isozymes in plant genetics and breeding, Part A”, Elsevier Science Publ., Amsterdam, pp. 381–400.
Arulsekar, S.; D.E. 1985 Parfitt & G.H. McGranahan. Isozyme gene markers in Juglans species. J. Hered. 76: 103–106.
Bergmann, F. 1973 Genetische Untersuchungen bei Picea abies mit Hilfe der Isoenzym-Identifizierung, II. Genetische Kontrolle von Esterase- und Leucinaminoceptidase-Isoenzymen im h a p l o i d e n Endosperm ruhender Samen. Theoretical and Applied Genetics 43: 222–225.
Bergmann, F. 1974 Genetic distance between populations. II. Determining the genetic distance b e t w e e n European populations of Picea abies on the basis of isozyme gene frequencies. Silvae Genet. 23: 28–32.
Bergmann, F. 1975 Adaptive acid phosphatase polymorphism in conifer seeds. Silvae Genetica 24: 5–6.
Bergmann, F. 1975a Herkunfts-Identifizierung von Forstsaatgut auf der Basis von Isoenzym-Genhäufigheiten. Allg. Forst- u. J.- Ztg. 146: 191–195.
Bergmann, F. 1978 The allelic distribution at an acid phosphatase locus in norway spruce (Picea abies) along similar climatic gradients. Theor. Appl. Genet. 52: 57–64.
Bergmann, F. 1987 Characterization of multiclonal aspen cultivars using isozyme electrophoresis. Forest Ecology Management 22: 167–172.
Bergmann, F. & R. Scholz. 1987 The impact of air pollution on the genetic structure of norway spruce. Silvae Genetica 36: 80–83.
Bousquet, J.; W.M. Cheliak & M. Lalonde. 1987 Allozyme variability in natural populations of green alder (Alnus crispa) in Quebec. Genome 29: 345–351.
Bousquet. J.; W.M. Cheliak & M. Lalonde. 1988 Allozyme variation within and among mature populations of speckeld alder (Alnus rugosa) and relationships with green alder (Alnus crispa). Amer. J. Bot. 75: 1678–1686.
Cheliak, W.M. & J.A. Pitel. 1984 Electrophoretic identification of clones in trembling aspen. Can. J. For. Res. 14: 740–743.
Farmer, R.E.; W.M. Cheliak; D.J. Perry; P. Knowles; J. Barrett & J. Pitel. 1988 Isozyme variation in balsam poplar along a latitudinal transect in northwestern Ontario. Can. J. For. Res. 18: 1078–1081.
Fernald, M.L. 1950 Gray's manual of botany. Eighth edition. American Book Comp.
Fineschi, S. 1988 Short Note. Genetics of Chestnut (Castanea sativa Mill.). Silvae Genetica 37: 82–83.
Fowler, D.R. & R.W. Morris. 1977 Genetic diversity in red pine: evidence for low genetic heterozygosity. Can. J. Forest Res. 7: 343–347.
Gerhold, H.D. 1977 Effect of air pollution on Pinus strobus L. and genetic resistance. A literature review. Corvallis Environ. Res. Lab., EPA 600/3–77–002.
Gregorius, H.-R. 1985 H.H. Hattemer, F. Bergmann, G. & Müller-Starck, Umweltweltbelastung und Anpassungsfähigkeit von Baumpopulationen. Silvae Genetica 34: 230–241.
Guzina, V. 1978 Genetic control of isoperoxidases Px-A1, Px-A2, Px-B1, Px-B2 in aspen (Populus tremula). Ann. Forestales 8: 51–90.
Houston, D. 1978 Genetic variation in peroxidase isozymes of Quercus rubra L. Fifth North American For. Biol. Workshop. Gainsville. Fla., March, 1978.
Hyun, O.J.; O.P. Rajora & L. Zsuffa. 1987 Genetic variation in trembling aspen in Ontario based on isozyme studies. Can. J. For. Res. 17: 1134–1138.
Kephart, S.R. 1990 Starch el electrophoresis of plant isozymes: A comparative analysis oftechniques. Amer. J. Bot. 77(5): 693–712.
Kim, Z.-S. 1979 Inheritance of Leucine Aminopeptidase and Acid Phosphatase Isozymes in Beech (Fagus sylvatica L.). Silvae Genetica 28: 68–71.
Kormuták, A.; F. Bencat; D. Rudin & R. Seyedyazdani. 1982 Isoenzyme variation in the four Slovakian populations of Abies alba Mill. Biologia (Bratislava) 37: 433–440.
Lagercrantz, U.; N. Ryman & G. Stähl. 1988 Protein loci in diploid tissue of norway spruce (Picea abies K.): description and interpretation of electrophoretic variability patterns. Hereditas 108: 149–158.
Lundquist, K. 1979 Allozyme frequency distributions in four Swedish populations of norway spruce (Picea abies K.). Hereditas 90: 127–143.
Manos, P.S. & D.E. Fairbrothers. 1987 Allozyme variation in populations of six Northeastern American Red Oaks (Fagacea: Quercus subg. Erythrobalanus). Systematic Botany 12: 365–373.
Markert, C.L.(Ed.). 1975 Isozymes. Vol 1–4, Academic Press, New York.
Moran, G.F.; J.C. Bell & A.C. Matheson. 1980 The genetic structure and levels of inbreeding in a Pinus radiata D. Don seed orchard. Silvae Gent. 29: 190–193.
Müller, G. 1976 A simple method of estimating rates of self-fertilization by analyzing isozymes in tree seeds. Silvae Genetica 25: 15–17.
Müller-Starck, G. 1985 Genetic differences between “tolerant” and “sensitive” beeches (Fagus sylvatica L.) in an environmentally stressed adult forest stand. Silvae Genetica 34: 241–246.
Muona, O.; R. Yazdani & G. Lindquist. 1987 Analysis of linkage in Picea abies. Hereditas 106: 31–36.
Nei, M. & A.K. Roychoudhury. 1974 Sampling variances of heterozygosity and genetic distance. Genetics 76: 379–390.
Olsson, U. 1975 Peroxydase isozymes in Quercus petraea and Quercus robur. Bot., Notiser 128: 401–411.
Perry, D.J. & P. Knowles. 1988 Allozyme variation in sugar maple at the northern limit of its range in Ontario. Canada, Can. J. For. Res. 19: 509–514.
Poulsen, H.; V. Simonsen & H. Wellendorf. 1983 The inheritance of six isoenzymes in norway spruce Picea abies (L.) Karst. Forest Tree Improvement 16: 13–33.
Rothe, G.M. & W.D. Maurer. 1986 One-dimensional PAA-gel electrophoretic techniques to separate functional and denatured proteins, in: M. Dunn (Ed.) Gel electrophoresis of proteins, Wright, Bristol.
Rothe, G.M. 1988 Möglichkeiten and Grenzen von Isozym-Analysen zur Ermittlung der genetischen Konstitution von Waldbäumen. Allg. Forst Z., 43(49): 1336–1338.
Rothe, G.M. 1990 Identifizierung von Fichtenklonen durch Isozymanalysen an Nadeln. Allg. Forst-u. J. Ztg. 161: 104–107.
Scholz, F. & F. Bergmann. 1984 Selection pressure by air pollution as studied by isozyme-gene-systems in norway spruce exposed to sulphur dioxide. Silvae Genetica 33: 238–240.
Shaw, D.V. & R.W. Allard. 1981 Analysis of mating system parameters and population structure in Douglas-fir using single-locus and multi-locus methods, in: Conkle, M.T. (Technical Coordinator), Proc. Symp. Isozymes North Am. forest trees and forest insects, Berkeley, C.A. USDA Forest Serv. Gen. Techn. Rep. PSW-48, pp. 18–22.
Shen, H.- H.; D. Rudin & D. Lindgren. 1981 Study of the pollination pattern in a Scots pine seedorchard by means of isozyme analysis. Silvae Genet. 29: 190–193.
Steinhoff, R.J.; D.G. Joyce & L. Fins. 1983 Isozyme variation in Pinus monticola. Can. J. For. Res. 13: 1122–1132.
Surles, S.; J.L. Hawrick & B.C. Bongarten. 1989 Allozyme variation in black locust (Robinia pseudoacacia). Can J. For. Res. 19: 471–479.
Tigerstedt, P.M.A. 1973 Studies on isozyme variation in marginal and central populations of Picea abies. Hereditas 75: 47–60.
Tobolski, J.J. 1978 Isozyme variation in several species of oaks. Proceedings of the Second Central Hardwood Forest Conference, Purdue University, West Lafayette, Indiana, November
Tzschacksch, O. & M. Weiss. 1972 Die Variation der SO2 Resistenz von Provenienzen der Baumart Fichte (Picea abies (L.)) Karst. Beitr. Forstwirtsch. 3: 21–23.
Weber, J.C. & R.F. Stettler. 1981 Isoenzyme variation among ten populations of Populus trichocarpa Toor et Gray in the Pacific Northwest. Silvae Genetica 30: 82–87.
Wendel, J.F. & C.R. Parks. 1985 Genetic diversity and population structure in Camellia japonica L. (Theaceae). Am. J. Bot. 72: 52–65.
Yeh, F.C. & D. O'Malley. 1980 Enzyme variations in natural populations of douglas-fir Pseudotsuga menziesii (Mirb.) Franco, from British Columbia, 1. Genetic variation patterns in coastal populations. Silvae Genet. 29: 83–92.
Yeh, F.C. & Y.A. El-Kassaby. 1989 Enzyme variation in natural populations of sitka spruce (Picea sitchensis), 1. Genetic variation patterns among trees from 10 IUFRO provenances. Can. J. For. Res. 10: 415–422.
les isoenzymes provenant de loci multiples, qui codent des chaînes polypeptidiques de l'enzyme structurellement distinctes;
les alléloenzymes (ou allozymes), qui sont des variantes structurellement distinctes d'une chaîne polypeptidique particulière, codées par des allèles multiples à un locus unique;
les isoenzymes secondaires, qui résultent de modifications de la structure de l'enzyme postérieures à la traduction génétique.
La distinction entre allèles multiples et loci multiples comme causes de la formation des isoenzymes réside dans le fait que les allèles multiples sont le résultat de différences entre individus d'une certaine espèce, tandis que les loci multiples sont communs à tous les individus d'une espèce.
La distance génétique (do) est la mesure chiffrée de la différence entre les distributions statistiques de fréquences de gènes. Elle peut varier de 0 à 1. Elle atteint la valeur l lorsqu'll n'y a pas d'allèles communs entre les deux populations. La valeur 0 indique que les populations sont génétiquement identiques (Rothe, 1988).
L'hétérozygotie moyenne est identique à la probabilité moyenne pour que deux gènes choisis au hasard nesoient pas identiques. Ce chiffre est indépendant de la fréquence des hétérozygotes observés, du fait qu'il ne prend pas en compte les croisements non aléatoires et la sélection. Il est basé uniquement sur les fréquences de gènes (Rothe, 1988).
5 Union internationale des instituts de recherche forestière.