Gunter M. Rothe
Institut für Allgemeine Botanik, Fachbereich Biologie
Johannes Gutenberg-Universität
Saarstr. 21, 6500 Mainz, Alemania
1. INTRODUCCION
Los estudios sobre genética de árboles forestales se han efectuado utilizando principalmente tres clases de rasgos simplemente heredados: los marcadores morfológicos, las variantes en monoterpenos y las isoenzimas.
Los rasgos morfológicos son fáciles de determinar pero su empleo en los estudios genéticos es limitado porque las características morfológicas fenotípicas están generalmente controladas por varios genes y los factores cuantitativos están sometidos a una fuerte influencia ambiental. En consecuencia, no se pueden identificar las contribuciones de genes individuales y no pueden detectarse diferencias genéticas específicas entre individuos.
En las gimnospermas, genes individuales controlan el nivel de unos pocos monoterpenos. Sin embargo, la interpretación genética de los patrones de monoterpenos es complicada y el análisis de un gran número de muestras, mediante cromatografía “gas líquido”, es muy costoso y lleva mucho tiempo.
En comparación con lo anterior, las isoenzimas son relativamente fáciles de manejar. Su empleo se introdujo en la genética de árboles forestales hace unos 15 años y en la actualidad está muy difundido basándose en la hipótesis de que las isoenzimas son productos directos de alelos específicos. Una vez identificadas, se dispone de métodos estadísticos para estudiar su distribución (frecuencias) inter e intrapoblacionalmente.
2. LAS ALOENZIMAS COMO MARCADORES GENETICOS
Los organismos diploides, que se reproducen sexualmente, reciben un juego completo de cromosomas de cada progenitor. Consecuentemente, tienen cada gen por duplicado; los genes homólogos se denominan alelos. Si existe más de un alelo en el locus de un gen estructural, las diversas enzimas determinadas por ellos pueden presentarse, ya sea en un organismo diploide simple (heterozigótico) o en distintos miembros (homozigóticos) de una especie.
El número de alelos en un locus dado depende de la especie y del propio locus. Los genes se caracterizan como polimórficos si comprenden dos o más alelos. Los genes invariantes (monomórficos) no son adecuados para estudios genéticos.
Para interpretar correctamente los patrones de isoenzimas2, debe conocerse la composición de sus subunidades (número de polipéptidos que constituyen la enzima completa) (Figura 1).
Los genes que codifican aloenzimas, se identifican mediante una abreviatura del nombre de la enzima, p.ej. “Aat”-para aspartato amino transferasa. Si un gen comprende más de un locus (el lugar de la molécula de ADN definido con precisión) estos loci se indican mediante una letra mayúscula (o un número) ligado al nombre del gen como un sufijo, p.ej. Aat-A o Aat-B (o Aat-1 o Aat-2). Los alelos correspondientes pueden indicarse mediante números consecutivos como Aat-A1, Aat-A2, etc. (o Aat-11, Aat-12) (Figuras 2, 3; Cuadros 1, 2).
Las aloenzimas se pueden emplear en estudios genéticos si se cumplen las siguientes condiciones:
el genotipo está expresado fenotípicamente mediante patrones de enzimas dados, que pueden observarse en el laboratorio. Un requisito previo para ello es que las formas enzimáticas de interés segreguen en forma mendeliana (como se muestra en la Figura 1); y
por lo menos un locus por enzima es polimórfico.
Se pueden identificar genotipos específicos empleando:
el examen de progenies resultantes de cruzamientos controlados;
estudios comparativos de tejidos haploides y diploides de un individuo (endosperma haploide y embrión diploide de las gimnospermas o polen haploide y embrión diploide de las angiospermas); y
aplicando la ley de Hardy-Weinberg en modelos teóricos (Rothe 1988).
2.2. INSTRUMENTOS PARA DETECTAR ISOENZIMAS
La identificación de isoenzimas se basa en su alta específícídad de sustrato y en sus variables movilidades electroforéticas. Si dos variantes alélicas de una enzima difieren en el número de aminoácidos provistos de carga, sus diferentes cargas netas se pueden emplear para separarlas mediante electroforesis. La estructura primaria (espina dorsal del péptido) de estas enzimas está codificada en el individuo en las dos hélices homólogas del ADN y, de acuerdo con ello, los genes alélicos se pueden identificar mediante alozimas con carga diferente. Sin embargo, lamentablemente dos tercios de los aminoácidos protéicos no están cargados, lo que reduce la posibilidad de identificación de variantes de las enzimas alélicas existentes, a menos del 30% del número total presente. No obstante, para aquellas variantes que tienen carga, los métodos electroforéticos son relativamente sencillos de realizar en la práctica. Es suficiente colocar extractos brutos de células o tejidos en algun tipo de medio de separación electroforética, como el gel de almidón -celo gel- o el gel de poliacrilamida; y subsiguientemente teñir el gel empleando un colorante que permita hacer visible sólo una enzima específica y sus formas moleculares múltiples, dejando las otras proteínas separadas (y enzimas) sin teñir (Rothe & Maurer 1986, Kephart 1990). Finalmente las formas teñidas de enzimas se cuentan y se utilizan las frecuencias calculadas para nuevos cálculos genéticos (véase la Sección 2. posterior; cf. también la Figura 2.3).
3. APLICACION DE LA INVESTIGACION DE ALOENZIMAS
3.1 IDENTIFICACION DE LOTES DE SEMILLA
Las diferencias en las frecuencias de aloenzimas se pueden emplear para determinar el origen de un lote de semillas o de brinzales procedentes de un huerto semillero o de diferentes poblaciones de una especie arbórea determinada (Adams 1983 y Bergmann 1975a. Véase también p.ej. Houston 1978, como ejemplo de identificación de seis fuentes geográficamente diferentes de semilla de Quercus rubra, mediante el empleo de perixodasas). Sin embargo, especialmente en Europa Central, es difícil la identificación de procedencias de abeto rojo de Noruega (Picea abies) porque se han introducido un gran número de procedencias y se desconoce su verdadero origen (Bergmann 1975a).
Para la identificación de procedencias de abeto rojo deben analizarse por lo menos 7 semillas de cada uno de 200 árboles, para determinar las frecuencias de aloenzimas (Adams 1983, Bergmann 1975a). De acuerdo con Bergmann (1975a), se encontraron en las semillas iguales frecuencias de aloenzimas aunque procedieran de árboles individuales de una cierta población o de una mezcla de semillas de tal población. El examen de 100 sondas de genoma diploide recogidos de un huerto semillero (6 50 endospermas haploides de una especie conífera) permite la detección de alelos que aparecen con una frecuencia de 0,03 o superior, con una probabilidad del 95% (Adams 1983). Sin embargo, si se sospecha la existencia de una seria contaminación de polen de fuentes extrañas al huerto semillero y se realiza un análisis de isoenzimas para demostrarlo, se recomienda duplicar la muestra anterior hasta 100 endospermas haploides de semillas (Adams 1983).
En muchos países los bosques han sido divididos geográfica, topográfica y ecológicamente en zonas de semilla. Se supone que cada zona representa una unidad ambiental relativamente homogénea, dentro de la cual los genotipos están adaptados, en principio, para su utilisación en cualquier otra parte de la misma zona, mientras que las semillas del exterior de ésta es de prever que no estén tan bien adaptadas (Adams 1983). Limitando la semilla utilizada para reforestación a la recolectada en el interior de las zonas de semilla seleccionadas, se confía en garantizar de este modo que la semilla esté debidamente adaptada a las condiciones del sitio de plantación. La identidad de los lotes de semilla puede verificarse siguiendo su recolección mediante el empleo de técnicas de isoenzimas. No obstante, si la diversidad genética entre poblaciones es pequeña (do <0, 10)3 se limitará el uso de los estudios de isoenzimas en la identificación de lotes de semilla procedentes de varias zonas de semilla (Adams 1983).
3.2 IDENTIFICACION DE CLONES
Las isoenzimas se pueden utilizar también para identificar árboles individuales estrechamente relacionados, o clones (Adams 1983). Los estudios de isoenzimas pueden realizarse con yemas invernales, hojas o acículas del año. Para la identificación de clones es suficiente comparar patrones de isoenzimas fenotípicas, incluso sin conocer su base genética (Cheliak & Pitel 1984 y Bergmann 1987). En un estudio, el análisis de 6 a 7 sistemas de enzimas variables, incluyendo de 10 a 20 loci diferentes, permitió la certificación de 50 a 100 clones de una variedad multiclonal del abeto rojo de Noruega (Picea abies) (véase el Cuadro 3). Esta aplicación de los estudios de isoenzimas tiene potencialmente gran importancia, porque en algunos países se han desarrollado variedades multiclonales como parte de una estrategia de selvicultura clonal para ayudar a evitar la reducción de la diversidad genética en generaciones futuras de poblaciones artificiales (Rothe 1990).
3.3 DETERMINACION DE LA FIABILIDAD DEL CRUZAMIENTO CONTROLADO Y NIVELES DE AUTOFERTILIZACION
La fiabilidad del cruzamiento controlado se puede evaluar examinando los patrones de isoenzimas en las semillas resultantes de tales cruzamientos, comparando los resultados con los previstos teóricamente, basados en los genotipos de los progenitores (Adams 1983). Una polinización controlada se puede considerar un fracaso si las semillas de una de cada 4 muestras (o una de cada dos para las especies coníferas) contienen alelos que no estén presentes en ninguno de los progenitores supuestos (Adams 1983). Los experimentos realizados con pino de Oregón (Pseudotsuga menziesii) demostraron que la mayoría de los fracasos en los cruzamientos controlados se deben a la contaminación de polen, por polen extraño (Adams 1983).
Además, se pueden utilizar los marcadores genéticos de isoenzimas para identificar árboles que se autofertilizan en los huertos semilleros (Müller 1976, Moran; Bell & Matheson 1980 y Shen; Rudin & Lindgren 1981). Como la autofertilización afecta a todo el genoma por igual, puede descubrirse mediante la investigación de un único locus polimórfico. En el abeto rojo de Noruega (Picea abies) para el locus B de la leucina aminopéptidasa (Lap-B) se produjo autofertilización en dos de cada cuatro genotipos. Se estimó que la tasa de autofertilización era del 15% (Müller 1976). Teniendo en cuenta que la diversidad genética de las semillas de árboles de autofertilización se reduce, generalmente conviene identificar y eliminar tales individuos de autofertilización, en los huertos semilleros (Shaw & Allard 1981).
3.4 IDENTIFICACION DE ESPECIES DE ARBOLES
La variación de los patrones de las aloenzimas puede utilizarse para distinguir distintas especies o híbridos de árboles. Las semillas de dos especies de alerce, Larix decidua y Larix kaempferi, se separaron mediante análisis de la movilidad de las enzimas sikimato-deshidrogenasas. En el L. kaempferi la enzima migró más rápidamente al ánodo que en el L. decidua. En híbridos de ambas especies parentales se pudieron identificar ambas variantes enzimáticas (Bergmann 1975a).
Los robles (Quercus spp.) comprenden un grupo taxonómicamente complejo. En los Estados Unidos existen más de 50 especies de robles de porte arbóreo y el número total de especies de este género pasa de 300. Algunas de éstas poseen rasgos morfológicos y anatómicos únicos y fácilmente identificables, mientras otras son difíciles de identificar y diferenciar entre sí. La identificación taxonómica se complica aún más por la hibridación aparente entre unas cuantas especies. Más de 60 híbridos han sido registrados sólo en los Estados Unidos (Fernald & Gray 1950).
Investigaciones realizadas con yemas y tejidos cotiledonales de bellota procedentes de diversas fuentes de semilla de roble rojo del norte (Quercus rubra L.), roble negro (Q. velutina Lam.), roble blanco (Q. alba L.), roble blanco de los pantanos (Q. bicolor Wild), roble “bur” (Q. macrocarpa Michx.) y roble de “chinkapin” (Q. muehlenbergii Engelm.), demostraron que la mayoría de las bandas isoenzimáticas de leucina aminopeptidasa, alcohol deshidrogenasa, ácido fosfatasa y aspartato aminotransferasa, eran comunes a dos o más de estas especies (Tobolski 1978). Esta semejanza en los patrones de isoenzimas es probable que refleje su estrecha relación taxonómica (y por tanto genética). Sin embargo, en el análisis, los individuos de algunas especies mostraron genotipos distintos; éstos pudieron distinguirse también con facilidad porque mostraban fenotipos diferentes (Tobolski 1978).
Las especies Quercus petraea y Quercus robur pudieron clasificarse de acuerdo con sus patrones de peroxidasa (Olsson 1975). Algunas poblaciones de Q. petraea mostraron pruebas evidentes de introgresión con el Q. robur.
3.5 CARACTERISTICAS DE LAS POBLACIONES DE ARBOLES FORESTALES
Además de los usos anteriores, se han estudiado las siguientes características genéticas de poblaciones empleando frecuencias de aloenzimas:
el nivel de variabilidad genética dentro de una población;
la variación entre poblaciones;
posibles factores implicados en la variación observada.
La estructura genética de las poblaciones, reflejada en la varianza “inter-locus” en los estudios de aloenzimas, está sujeta a fuerzas evolutivas tales como la mutación, la selección y la deriva genética. La varianza “intra-locus”, identificable en los estudios de laboratorio, depende del tamaño de la muestra y de las frecuencias de los genes en el locus estudiado (Nei & Roychoudhury 1974). El grado de heterozigosis4 (H) en las poblaciones de árboles forestales se ha encontrado que varía de 0,19 a 0,41, siendo típicos de las coníferas los valores más elevados (Cuadros 4 y 5).
La variación alélica se puede definir como “la proporción prevista de heterozigotos por locus y por población”, y se suele indicar con una h. Los cálculos del valor medio de h de 10 poblaciones distintas de Picea sitchensis (Boug. Carr.), importante árbol forestal originario de la costa occidental de Norteamérica-, dieron valores que oscilaban entre 0,475 (glucosa- 6-fosfato deshidrogenasa, G6pdh) a 0,001 (fosfoglucomutasa, Pgm2). Este amplio rango de h, con valores próximos a 0, es característico de varias especies coníferas (Yeh & El-Kassaby 1980).
El nivel de diversidad genética de las coníferas suele ser alto. Esto se debe a: (i) una selección divergente para su adaptación macrogeográfica, (ii) una selección equilibrada para su diferenciación microgeográfica y (iii) un sistema de mejora genética que facilita el flujo de genes dentro de subpoblaciones y entre ellas. La heterosis puede favorecer aún más el mantenimiento de la variabilidad genética (Yeh & El-Kassaby 1980). Los estudios realizados sobre variación genética en diez poblaciones de IUFRO5 de abeto rojo de sitka (Picea sitchensis) demonstraron que (i) la amplitud de polimorfismo genético variaba considerablemente de un locus a otro; (ii) varias poblaciones eran similares en cuanto a la amplitud y patrón de variabilidad genética, respecto a la mayoría de los loci estudiados; en tanto que (iii) algunos loci específicos mostraron grandes diferencias entre poblaciones (Yeh & El-Kassaby 1980). A pesar de las grandes distancias geográficas entre los orígenes de las semillas ensayadas, las diferencias de constitución genética del abeto rojo de sitka no aumentaron apreciablemente en proporción con la distancia geográfica.
Las diferencias genéticas entre poblaciones las exponen de diferente forma a la selección natural, a nivel de población, favoreciendo la selección de la población (Yeh & El-Kassaby 1980). Esta conclusión se ha confirmado para varias especies arbóreas, además del abeto rojo de sitka, como por ejemplo la Picea abies (L.) Karst. (Bergmann 1974), Pinus monticula Dougl. (Steinhoff; Joyce & Fins 1983) y Pseudotsuga menziesii (Mirb.) Franco (Yeh & O'Malley 1980). En Escandinavia las frecuencias de los alelos situados en el locus para el ácido fosfatasa B eran más frecuentes en las poblaciones meridionales del abeto rojo de Noruega que en las nórdicas (Bergmann 1978).
Generalmente las coníferas presentan altos niveles de heterozigosis. Una excepción notable a esta regla es el Pinus resinosa (Fowler & Morris 1977). Una de las teorías que explican la falta de variabilidad del P. resinosa es que ésta es el resultado de un cuello de botella importante en la población que se produjo probablemente durante el período Pleistoceno, cuando la distribución de la especie se redujo a pequeñas poblaciones aisladas, como resultado de la glaciación.
La variabilidad genética de los árboles caducifolios es generalmente menor que en las coníferas. En 17 poblaciones de Quercus, el valor medio de los loci polimórficos era del 30%, con 2,32 alelos por locus polimórfico. Unas estimaciones mínimas de diversidad genética entre poblaciones pueden indicar que hay mayor variabilidad genética dentro de las poblaciones que entre unas y otras de éstas (Manos & Fairbrothers 1987).
3.6 ADAPTACION DE LOS ARBOLES A DIFERENTES AMBIENTES
Los árboles son organismos de vida prolongada y por ello, deben estar bien adaptados a sus hábitats originales y a las fluctuaciones ambientales previstas en tales hábitats para largos períodos de tiempo. Esto se puede conseguir, ya sea mediante una extensa adaptación dentro de la población o mediante adaptaciones específicas de la población a las condiciones específicas de la estación. En el abeto rojo de Noruega (Picea abies) y en el pino de Oregon (Pseudosutga menziesii), se identificó un gen de un locus que codifica la fosfatasa ácida, que mostró una variación importante dependiente del medio ambiente. En ambas especies los alelos Acp- que codifican enzimas con una banda única dominan en las zonas más frías del ámbito de distribución de la especie, mientras que los alelos que codifican enzimas de dos bandas se dan con más frecuencia en todas las zonas climáticas moderadas (Bergmann 1975).
La variación entre poblaciones se puede cuantificar mediante un número sencillo, la distancia genética (d0) (véase p. ej. Nei & Roychoudhury 1974 y Surles; Hawrick & Bongarten 1989). Unas poblaciones completamente diferentes genéticamente se caracterizan por un valor d0 = 1, mientras que el valor 0 corresponde a poblaciones idénticas. En rodales finlandeses de Picea abies, se ha observado una distribución totalmente al azar de los genes dentro de los rodales, utilizando aloenzimas de leucina aminopeptidasa, Lap.; peroxidasa, Pod y esterasa, Est. (Tigerstedt 1973). Sin embargo, para otra serie de especies parece que se da una correlación entre las distancias genéticas y las geográficas, p.ej. la Camellia japonica L. (Wendel & Parks 1985), Picea abies (L) Karst. (Bergamnn 1975), Pinus monticola Dougl. (Steinhoff; Joyce & Fins 1983) y Pseudotsuga menziesii (Mirb.) Franco (Yeh & O'Malley 1980). El aislamiento debido a la distancia puede ser un factor importante en la diferenciación de las poblaciones de estas especies (cf. Perry & Knowles 1988).
3.7 IMPACTO DE LOS CONTAMINANTES DEL AIRE EN LA ESTRUCTURA GENETICA DE LOS ARBOLES FORESTALES
Numerosos datos referentes a la diferenciación genética de los árboles respecto a su sensibilidad a los contaminantes, han sido examinados por Gerhold (1977). Estadísticamente, existen diferencias significativas entre poblaciones del abeto rojo de Noruega (Picea abies) respecto a su sensibilidad al SO2. Las poblaciones más tolerantes parecen ser las de origen septentrional y elevada altitud (Tzschacksch & Weiss 1972). El grado medio de heterozigosis de individuos de abeto rojo que proceden de las montañas del Harz, en Alemania, que no se dañaban cuando se exponían al SO2 y al O3, era mayor que la de los individuos dañados (Bergmann & Scholz 1987). Sin embargo, las procedencias de abeto rojo de Noruega tolerantes al SO2 mostraron menores frecuencias de alelos raros, situados en los loci Gpdh-A1 y Mdh-C3, que las subpoblaciones que eran sensibles al SO2 (Bergmann & Scholz 1987). En contraste con las complejas variables ambientales, los contaminantes simples del aire, como el SO2 o el O3, conducen a una selección tendente a una diversidad genética reducida (Bergmann & Scholz 1987). Observaciones similares se hicieron para el haya (Fagus sylvatica) en la cual la selección de genotipos resistentes tuvo lugar sobre todo en la primera (F1) generación (Müller-Starck 1985).
Dos conclusiones principales pueden deducirse de las investigaciones sobre el impacto de los contaminantes del aire en la estructura genética de los árboles forestales:
los árboles fuertemente heterozigóticos parecen estar mejor adaptados, a una multiplicidad de impactos ambientales que actúan simultáneamente, que aquellos árboles que tienen un grado menor de heterozigosis, y
la polución de contaminantes del aire ocasiona una reducción de la diversidad genética de la población. Las razones de esta disminución de la diversidad (y, por lo tanto, de la adaptabilidad general) son (1) los mayores niveles de selección natural direccional y (2) la disminución del flujo de genes.
La resistencia de los árboles forestales a los contaminantes del aire se supone que está determinada por genes preadaptativos. Estos son genes muy adaptativos que existen ya en el genoma pero que no son identificables antes de su exposición al agente tóxico (Gregorius; Hattemer; Bergmann & Müller-Starck 1985).
Nota del Editor: Para información adicional sobre biotecnología en programas de mejoramiento de árboles, véase también el artículo de Cheliak, W.M. & Rogers, D.L. en la sección (iv) de la Bibliografía Reciente de Interés.
Figura 1
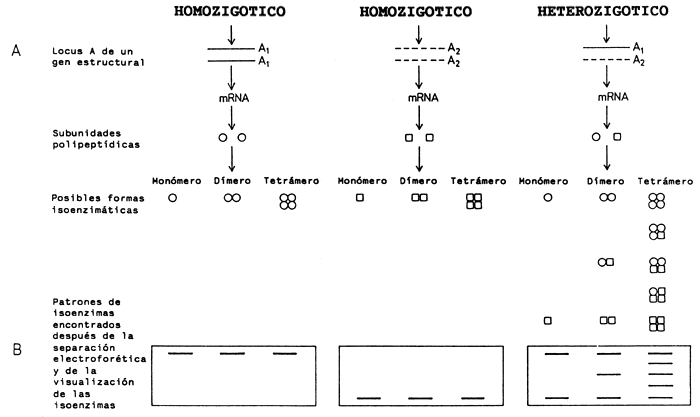
Representación esquemática de los patrones de isoenzimas resultantes en heterozigotos cuando la enzima es un monómero, dímero o tetrámero (que contiene uno, dos o cuatro polipéptidos) y aparecen por lo menos dos alelos diferentes en el locus que los codifica. Se supone además que las subunidades combinan libremente y segregan de acuerdo con las pautas mendelianas.
Figura 2
Quercus robur L.
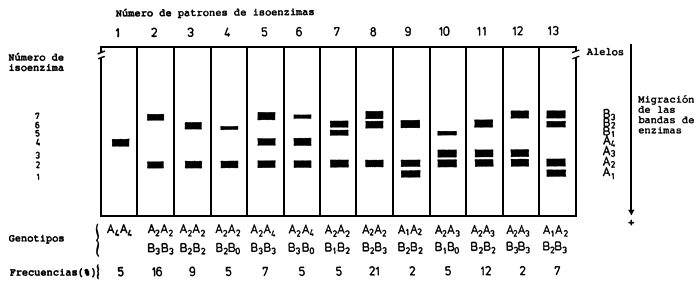
Patrones de isoenzimas e interpretación genética de la leucina aminopeptidasa, encontrados en Quercus robur. Las isoenzimas fueron extraídas de 35 tejidos cotiledonales de bellotas de Quercus robur, separadas mediante electroforesis en gradiente sobre gel de poliacrilamida y finalmente visualizadas dentro del gel mediante aplicación de un método de teñido histoquímico. Las bellotas procedían de un rodal de Q. robur de Rheiland Pfalz (Alemania). Se observó un total de 13 patrones distintos de isoenzimas, comprendiendo 7 isoenzimas diferentes. Se supone que dos loci diferentes (Lap-A y Lap-B) codifican estas siete isoenzimas, que representan cuatro alelos en el locus A y tres alelos en el locus B (Dankwardt y Rothe, sin publicar).
Figura 3
Quercus petraea (Matt.) Liebl.
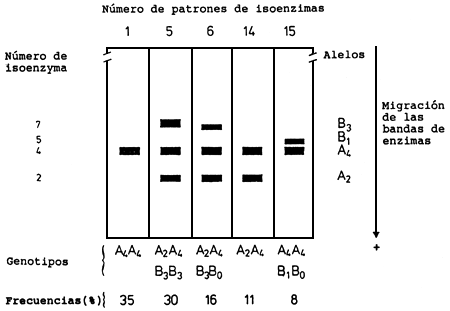
Patrones de isoenzimas observados en 35 tejidos cotiledonales de bellotas de Quercus petraea. Las bellotas procedían de un rodal de Rheiland Pfalz (Alemania). En comparación con el Quercus robur (Figura 2) solo se identificaron 5 en vez de 13 patrones de isoenzimas. Los patrones 1, 5 y 6 son similares a los patrones de los mismos números del Quercus robur pero difieren las frecuencias. Los patrones 14 y 15 se encontraron exclusivamente en Q. petraea mientras que los patrones de isoenzimas 2, 4 y 7–13 eran característicos del Q. robur. De acuerdo con ello, las poblaciones de las especies pueden distinguirse por sus patrones de isoenzimas. No obstante, la identificación de individuos de ambas especies requiere nuevos estudios sobre sistemas de isoenzimas (Dankwardt y Rothe, sin publicar).
Cuadro 1
Loci de genes enzimáticos y número de alelos encontrados en el abeto rojo de Noruega (Picea abies (L.) Karst.) y empleados para estudios genéticos
| Aspartato aminotransferasa | Aat-A | Aat-B | Aat-C | ||
| 3 | 3 | 1 | Lagercrantz 1988 | ||
| 4 | 4 | 4 | Lundquist 1979 | ||
| 2 | 1 | 3 | Poulsen et al. 1983 | ||
| 2 | 3 | Muona et al. 1987 | |||
| Fosfatasa ácida | Acp-A | Acp-B | |||
| 1 | 4 | Bergmann 1978 | |||
| 7 | Lunquist 1977 | ||||
| Diaforasa | Dia-A | Dia-B | |||
| 2 | 1 | Lagercrantz 1988 | |||
| Esterasa | Est-A | Est-B | Est-C | ||
| 3 | 3 | 2 | Bergmann 1973 | ||
| Glucosa-6 fosfato deshidrogenasa | Gpdh-A | ||||
| 2 | Scholz & Bergmann 1984 | ||||
| 3 | Muona et al. 1987 | ||||
| Glutamato deshidrogenasa | Gdh-A | Gdh-B | |||
| 2 | Scholz & Bergmann 1984 | ||||
| 2 | Lundquist 1979 | ||||
| 2 | Lagercrantz 1988 | ||||
| Glucosa-6 fosfato deshidrogenasa | Gpi-A | Gpi-B | |||
| 5 | 1 | Lagercrantz 1988 | |||
| Isocitrato deshidrogenasa | Idh-A | Idh-B | |||
| 2 | 2 | Scholz & Bergmann 1984 | |||
| 3 | 1 | Lagercrantz 1988 | |||
| Leucina aminopeptidasa | Lap-A | Lap-B | |||
| 1 | 4 | Bergmann 1973 | |||
| 4 | 4 | Lundquist 1979 | |||
| 3 | 4 | Poulsen et al. 1983 | |||
| 5 | 5 | Lagercrantz 1988 | |||
| Malato deshidrogenasa | Mdh-A | Mdh-B | Mdh-C | Mdh-D | |
| 1 | 2 | 2 | 2 | Poulsen et al. 1983 | |
| 4 | Scholz & Bergmann 1984 | ||||
| ? | 2 | 1 | ? | Lagercrantz 1988 | |
| Fosfoglucosa isomerasa | Pgi-A | Pgi-B | |||
| ? | 4 | Muona et al. 1987 | |||
| 6-Fosfogluconato deshidrogenasa | Pgd-A | Pgd-B | Pgd-C | ||
| 2 | 4 | 3 | Poulsen et al. 1983 | ||
| 4 | 1 | 1 | Lagercrantz 1988 | ||
| Fosfoglucomutasa | Pgm-A | Pgm-B | |||
| 2 | 2 | Poulsen et al. 1983 | |||
| 2 | 2 | Scholz & Bergmann 1984 | |||
| Superoxido dismutasa | Sod-A | Sod-B | |||
| 1 | 1 | Lagercrantz 1988 |
Cuadro 2
Loci de genes enzimáticos y alelos estudiados en árboles caducifolios y utilizados para análisis genéticos
| Especies | Loci de enzimas y número de alelos | Referencias | ||||||||||||||||||||||||||||||||||
| Aat- | Aco- | Acp- | Ald- | Est- | Gdh | Gpdh- | Idh- | Lap- | Mdh- | Per- | Pgd- | Pgi- | Pgm- | |||||||||||||||||||||||
| A | B | C | A | B | A | B | A | B | A | B | A | B | A | B | A | B | A | B | C | A | B | C | D | A | B | C | D | A | B | C | A | B | C | |||
| Alnus crispa | 1 | 1 | 2 | 2 | 1 | 1 | 1 | 1 | 2 | ? | 2 | 3 | 1 | 2 | 3 | 1 | Bousquet et al. 1987 | |||||||||||||||||||
| Alnus rugosa | ? | 2 | 2 | 1 | 1 | 1 | 1 | 1 | 4 | 2 | 4 | 3 | 1 | 2 | Bousquet et al. 1987 | |||||||||||||||||||||
| Castanea sativa | 2 | 3 | 2 | Fineschi 1988 | ||||||||||||||||||||||||||||||||
| Fagus sylvatica | 2 | 4 | 4 | 2 (3) | 2 | 4 | 4 | 4 | ? | 3 | 2 | 2 (4) | 3 | 2 | Kim 1979, Müller starck 1985 | |||||||||||||||||||||
| Juglans regia | 2 | 2 | 1 | ? | 2 | 1 | 2 | 3 | 1 | Arulsekar et al. 1985 | ||||||||||||||||||||||||||
| Populus balsamifera | 3 | 2 | 2 | 2 | 2 | 1 | 3 | 1 | 3 | 1 | 4 | Farmer et al. 1988 | ||||||||||||||||||||||||
| Populus tremula | 3 | 2 | 1 | 2 | 2 | 2 | Guzina 1978, Bergmann 1987 | |||||||||||||||||||||||||||||
| Populus tremuloides | 1 | 1 | 3 | 1 | 2 | 3 | 2 | 3 | 4 | 2 | 3 | 3 | 4 | 2 | 2 | 3 | 4 | 3 | 3 | 4 | Hyun et al. 1987 | |||||||||||||||
| (4) | (3) | (1 3) | Cheliak y Pitel 1984 | |||||||||||||||||||||||||||||||||
| Populus trichocarpa | 2 | 1 | 4 | 3 | 1 | 2 | 1 | 2 | 2 | 1 | 2 | 1 | 2 | Weber y Stettler 1981 | ||||||||||||||||||||||
| Robinia pseudoacacia | 1 | 3 | 3 | 3 | 3 | 4 | 5 | 4 | 3 | 4 | 3 | 3 | 3 | 3 | 3 | 2 | 3 | 4 | 3 | Surles et al. 1988 | ||||||||||||||||
| Quercus petraea | 1 (2) | 2 | 2 | 1 | 3 | 3 | 9 | 4 | Dankwardt y Rothe unpubl. Aas 1987 | |||||||||||||||||||||||||||
| Quercus robur | 1 (2) | 2 | 2 | 1 | 3 | 4 | 9 | 4 | Dankwardt y Rothe unpubl. Aas 1987 | |||||||||||||||||||||||||||
| Quercus subg. Erythrobalanus | ? | 4 | 3 | ? | ? | 2 | 3 | 1 | 3 | 1 | 2 | ? | 8 | 5 | 4 | Manos y Fairbrothers 1987 | ||||||||||||||||||||
| Acrónimos | ||
| Aat | : | aspartato aminotransferasa |
| Aco | : | aconitasa |
| Acp | : | fosfatasa ácida |
| Ald | : | Aldolasa |
| Est | : | esterasa |
| Gdh | : | glucamato deshidrogenasa |
| Gpdh | : | glucosa-6-fosfato deshidrogenasa |
| Idh | : | isocitrato deshidrogenasa |
| Lap | : | leucina aminopeptidasa |
| Mdh | : | malato deshidrogenasa |
| Per | : | peroxidasa |
| Pdg | : | 6-fosfogluconato deshidrogenasa |
| Pgi | : | fosfoglucosa isomerasa |
| Pgm | : | fosfoglucomutasa |
Cuadro 3
Caracterización de 16 clones de abeto rojo de Noruega de injerto de yema tardío (Picea abies [L.] Karst.) empleando patrones de isoenzimas de seis diferentes enzimas de acículas del año (Rothe 1990)
| Número del clon: | 1G | 7G | 8G | 10G | 14G | 15G | 16G | 17G | 18G | 21G | 84B | 89B | 93B | 94B | 95B | 105B |
| Patrones fenotípicos de isoenzimas | ||||||||||||||||
| ADH | 1 | 2 | 7 | 3 | 7 | 5 | 3 | 5 | 7 | 5 | 8 | 8 | 1 | 6 | 4 | 8 |
| EST | 4 | 4 | 5 | 5 | 5 | 5 | 2 | 1 | 2 | 3 | 1 | 1 | 1 | 1 | 1 | 1 |
| G6pDH | 3 | 1 | 1 | 3 | 3 | 3 | 2 | 3 | 3 | 3 | 3 | 3 | 6 | 4 | 5 | 4 |
| LDH | 1 | 1 | 2 | 1 | 2 | 2 | 1 | 1 | 1 | 1 | 2 | 2 | 1 | 2 | 2 | 1 |
| 6PGDH | 3 | 1 | 4 | 4 | 2 | 2 | 3 | 5 | 5 | 6 | 3 | 3 | 3 | 7 | 5 | 3 |
| SHDH | 4 | 4 | 3 | 4 | 3 | 3 | 1 | 1 | 2 | 2 | 3 | 3 | 2 | 2 | 2 | 3 |
| Acrónimos : | ADH: alcohol deshidrogenasa; EST: esterasa (teñida con α-naftilfosfato y azul rápido RR); G6pDH: glucosa-6- fosfato deshidrogenasa; LDH: lactato deshidrogenasa; 6PGDH: 6-fosfogluconato deshidrogenasa; SHDH: Sikimato deshidrogenasa |
| NB: | Se eligieron los clones números 1 a 24 de árboles de la procedencia Goldap (G) mientras que los numerados del 84 al 105 se eligieron de abeto rojo de la procedencia Borken (B), ambos localizados en Polonia. |
Cuadro 4
Grado medio de heterozigosis (H) de algunas poblaciones de árboles caducifolios (cf. Perry y Knowles 1988)
| Especies | H |
| Acer saccharum | 0.27 |
| Alnus crispa | 0.136 |
| Populus balsamifera | 0.037 |
| Populus tremuloides | 0.42 |
| Populus trichocarpa | 0.09 |
| Quercus subg. Erythrobalanus | 0.081 |
Cuadro 5
Variabilidad genética de poblaciones de coníferas, expresada por su grado medio de heterozigosis (H) (cf. Kormutak et al. 1982)
| Especies | H |
| Pinus contorta | 0.19 |
| Pinus jeffreyi | 0.26 |
| Pinus lambertina | 0.26 |
| Pinus silvestris | 0.41 |
| Pinus taeda | 0.34 |
| Pseudotsuga menziesii | 0.33 |
| Picea sitchensis | 0.15 |
REFERENCIAS
Aas, G. 1988 Untersuchung zur Trennung und Kreuzbarkeit von Stiel- und Traubeneiche (Quercus robur L. und Q. petraea (Matt.) Liebl.), Inaugural-Dissertation zur Erlangung der Doktorwürde der Forstwissenschaftlichen Fakultät der Ludwig-Maximilians Universität, München.
Adams, W.T. 1983 Application of isozymes in tree breeding, in: Tanksley, S.D. and T.J. Orton (Eds.): “Isozymes in plant genetics and breeding, Part A”, Elsevier Science Publ., Amsterdam, pp. 381–400.
Arulsekar, S.; D.E. Parfitt & G.H. McGranahan. 1985 Isozyme gene markers in Juglans species. J. Hered. 76: 103–106.
Bergmann, F. 1973 Genetische Untersuchungen bei Picea abies mit Hilfe der Isoenzym-Identifizierung, II. Genetische Kontrolle von Esterase- und Leucinaminopeptidase-Isoenzymen im haploiden Endosperm ruhender Samen. Theoretical and Applied Genetics 43: 222–225.
Bergmann, F. 1974 Genetic distance between populations. II. Determining the genetic distance between European populations of Picea abies on the basis of isozyme gene frequencies. Silvae Genet. 23: 28–32.
Bergmann, F. 1975 Adaptive acid phosphatase polymorphism in conifer seeds. Silvae Genetica 24: 5–6.
Bergmann, F. 1975a Herkunfts-Identifizierung von Forstsaatgut auf der Basis von Isoenzym-Genhäufigheiten. Allg. Forst- u. J.- Ztg. 146: 191–195.
Bergmann, F. 1978 The allelic distribution at an acid phosphatase locus in norway spruce (Picea abies) along similar climatic gradients. Theor. Appl. Genet. 52: 57–64.
Bergmann, F. 1987 Characterization of multiclonal aspen cultivars using isozyme electrophoresis. Forest Ecology Management 22: 167–172.
Bergmann, F. & R. Scholz. 1987 The impact of air pollution on the genetic structure of norway spruce. Silvae Genetica 36: 80–83.
Bousquet, J.; W.M. Cheliak & M. Lalonde. 1987 Allozyme variability in natural populations of green alder (Alnus crispa) in Quebec. Genome 29: 345–351.
Bousquet, J.; W.M. Cheliak & M. Lalonde. 1988 Allozyme variation within and among mature populations of speckeld alder (Alnus rugosa) and relationships with green alder (Alnus crispa). Amer. J. Bot. 75: 1678–1686.
Cheliak, W.M. & J.A. Pitel. 1984 Electrophoretic identification of clones in trembling aspen. Can. J. For. Res. 14: 740–743.
Farmer, R.E.; W.M. Cheliak; D.J. Perry; P. Knowles; J. Barrett & J. Pitel. 1988 Isozyme variation in balsam poplar along a latitudinal transect in northwestern Ontario. Can. J. For. Res. 18: 1078–1081.
Fernald, M.L. 1950 Gray's manual of botany. Eighth edition. American Book Comp.
Fineschi, S. 1988 Short Note. Genetics of Chestnut (Castanea sativa Mill.). Silvae Genetica 37: 82–83.
Fowler, D.P. & R.W. Morris. 1977 Genetic diversity in red pine: evidence for low genetic heterozygosity. Can. J. Forest Res. 7: 343–347.
Gerhold, H.D. 1977 Effect of air pollution on Pinus strobus L. and genetic resistance. A literature review. Corvallis Environ. Res. Lab., EPA 600/3–77–002.
Gregorius, H.-R. H.H. Hattemer, F. Bergmann, G. & Müller-Starck, 1985 Umweltweltbelastung und Anpassungsfähigkeit von Baumpopulationen. Silvae Genetica 34: 230–241.
Guzina, V. 1978 Genetic control of isoperoxidases Px-A1, Px-A2, Px-B1, Px-B2 in aspen (Populus tremula). Ann. Forestales 8: 51–90.
Houston, D. 1978 Genetic variation in peroxidase isozymes of Quercus rubra L. Fifth North American For. Biol. Workshop. Gainsville. Fla., March, 1978.
Hyun, O.J.; O.P. Rajora & L. Zsuffa. 1987 Genetic variation in trembling aspen in Ontario based on isozyme studies. Can. J. For. Res. 17: 1134–1138.
Kephart, S.R. 1990 Starch gel electrophoresis of plant isozymes: A comparative analysis oftechniques. Amer. J. Bot. 77(5): 593–712.
Kim, Z.-S. 1979 Inheritance of Leucine Aminopeptidase and Acid Phosphatase Isozymes in Beech (Fagus sylvatica L.). Silvae Genetica 28: 68–71.
Kormuták, A.; F. Bencat; D. Rudin & R. Seyedyazdani. 1982 Isoenzyme variation in the four Slovakian populations of Abies alba Mill. Biologia (Bratislava) 37: 433–440.
Lagercrantz, U.; N. Ryman & G Stähl. 1988 Protein loci in diploid tissue of norway spruce (Picea abies K.): description and interpretation of electrophoretic variability patterns. Hereditas 108: 149–158.
Lundquist, K. 1979 Allozyme frequency distributions in for Swedish populations of norway spruce (Picea abies K.). Hereditas 90: 127–143.
Manos, P.S. & D.E. Fairbrothers. 1987 Allozyme variation in populations of six Northeastern American Red Oaks (Fagacea: Quercus subg. Erythrobalanus). Systematic Botany 12: 365–373.
Markert, C.L. (Ed.). 1975 Isozymes. Vol 1–4, Academic Press, New York.
Moran, G.F.; J.C. Bell & A.C. Matheson. 1980 The genetic structure and levels of inbreeding in a Pinus radiata D. Don seed orchard. Silvae Gent. 29: 190–193.
Müller, G. 1976 A simple method of estimating rates of self-fertilization by analyzing isozymes in tree seeds. Silvae Genetica 25: 15–17.
Müller-Starck, G. 1985 Genetic differences between “tolerant” and “sensitive” beeches (Fagus sylvatica L.) in an environmentally stressed adult forest stand. Silvae Genetica 34: 241–246.
Muona, O.; R. Yazdani & G. Lindquist. 1987 Analysis of linkage in Picea abies. Hereditas 106: 31–36.
Nei, M. & A.K. Roychoudhury. 1974 Sampling variances of heterozygosity and genetic distance. Genetics 76: 379–390.
Olsson, U. 1975 Peroxydase isozymes in Quercus petraea and Quercus robur. Bot., Notiser 128: 401–411.
Perry, D.J. & P. Knowles. 1988 Allozyme variation in sugar maple at the northern limit of its range in Ontario. Canada, Can. J. For. Res. 19: 509–514.
Poulsen, H.; V. Simonsen & H. Wellendorf. 1983 The inheritance of six isoenzymes in norway spruce Picea abies (L.) Karst. Forest Tree Improvement 16: 13–33.
Rothe, G.M. & W.D. Maurer. 1986 One-dimensional PAA-gel electrophoretic techniques to separate functional and denatured proteins, in: M. Dunn (Ed.) Gel electrophoresis of proteins, Wright, Bristol.
Rothe, G.M. 1988 Möglichkeiten und Grenzen von Isozym-Analysen zur Ermittlung der genetischen Konstitution von Waldbäumen. Allg. Forst Z., 43(49): 1336–1338.
Rothe, G.M. 1990 Identifizierung von Fichtenklonen durch Isozymanalysen an Nadeln. Allg. Forst- u. J. Ztg. 161: 104–107.
Scholz, F. & F. Bergmann. 1984 Selection pressure by air pollution as studied by isozyme-gene-systems in norway spruce exposed to sulphur dioxide. Silvae Genetica 33: 238–240.
Shaw, D.V. & R.W. Allard. 1981 Analysis of mating system parameters and population structure in Douglas-fir using single-locus and multi-locus methods, in: Conkle, M.T. (Technical Coordinator), Proc. Symp. Isozymes North Am. forest trees and forest insects, Berkeley, C.A. USDA Forest Serv. Gen. Techn. Rep. PSW-48, pp. 18–22.
Shen, H. - H. D. Rudin & D. Lindgren. 1981 Study of the pollination pattern in a Scots pine seedorchard by means of isozyme analysis. Silvae Genet. 29: 190–193.
Steinhoff, R.J.; D.G. Joyce & L. Fins. 1983 Isozyme variation in Pinus monticola. Can. J. For. Res. 13: 1122–1132.
Surles, S.; J.L. Hawrick & B.C. Bongarten. 1989 Allozyme variation in black locust (Robinia pseudoacacia). Can J. For. Res. 19: 471–479.
Tigerstedt, P.M.A. 1973 Studies on isozyme variation in marginal and central populations of Picea abies. Hereditas 75: 47–60.
Tobolski, J.J. 1978 Isozyme variation in several species of oaks. Proceedings of the Second Central Hardwood Forest Conference, Purdue University, West Lafayette, Indiana, November
Tzschacksch, O. & M. Weiss. 1972 Die Variation der SO2 Resistenz von Provenienzen der Baumart Fichte (Picea abies (L.)) Karst. Beitr. Forstwirtsch. 3: 21–23.
Weber, J.C. & R.F. Stettler. 1981 Isoenzyme variation among ten populations of Populus trichocarpa Toor et Gray in the Pacific Northwest. Silvae Genetica 30: 82–87.
Wendel, J.F. & C.R. Parks. 1985 Genetic diversity and population structure in Camellia japonica L. (Theaceae). Am. J. Bot. 72: 52–65.
Yeh, F.C. & D. O'Malley. 1980 Enzyme variations in natural populations of douglas-fir Pseudotsuga menziesii (Mirb.) Franco, from British Columbia, 1. Genetic variation patterns in coastal populations. Silvae Genet. 29: 83–92.
Yeh, F.C. & Y.A. El-Kassaby. 1989 Enzyme variation in natural populations of sitka spruce (Picea sitchensis), 1. Genetic variation patterns among trees from 10 IUFRO provenances. Can. J. For. Res. 10: 415–422.
El término general “isoenzimas” por definición, incluye un rango de tipos de enzimas con igual especificidad de sustrato, que se produce ya sea en un individuo o en distintos miembros de la misma especie (cf. Markert 1975). Las isoenzimas se dividen en tres clases dependiendo de la forma en que se biosintetizan:
las isoenzimas proceden de loci de genes múltiples, que codifican cadenas de polipéptidos de la enzima estructuralmente diferentes;
las aloenzimas (o aleloenzimas) son variantes estructuralmente diferentes de una cadena particular de polipéptidos, codificada por alelos múltiples de un único locus;
isoenzimas secundarias que proceden de modificaciones de la estructura de la enzima posteriores al proceso de “traducción”.
La distinción entre alelos múltiples y loci de genes múltiples como causas de formación de isoenzimas es que los alelos múltiples son el resultado de diferencias entre miembros individuales de una cierta especie, mientras que los loci múltiples son comunes a todos los miembros de una especie.
La distancia genética (do) es una cuantificación de la diferencia entre dos distribuciones estadísticas de frecuencias de genes. La distancia genética puede obtener valores que varían de 0 a 1. El valor 1 se obtiene cuando no se presentan alelos comunes en las dos poblaciones. El valor 0 indica, que las poblaciones son genéticamente idénticas (Rothe 1988).
El grado medio de heterozigosis es igual a la probabilidad media de que dos genes elegidos al azar no sean idénticos. La cifra es independiente de la frecuencia de los heterozigotos observados ya que no tiene en cuenta los cruzamientos no realizados al azar ni la selección. Se basa únicamente en las frecuencias de genes observadas. (Rothe 1988).
5 Unión Internacional de Organizaciones de Investigación Forestal.